工欲善其事 必先利其器
前面我们介绍了华大 DNBelab C SeriesTM 单细胞转录组定量的基本流程: DNBC4tools—华大DNBelab系列单细胞分析pipeline
明确需求其中在准备样本数据步骤有提到,多样本处理首先需要制作一个自己的样本信息对应列表sample.tsv :
比如我需要处理的样本文件名是:
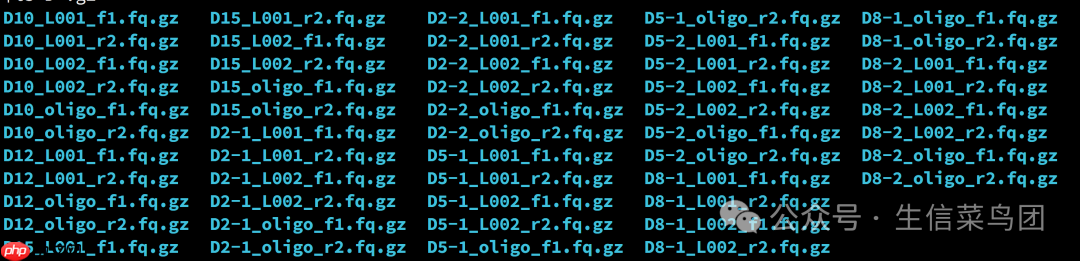 图片
图片需要生成的sample.tsv 文件格式是:
<code class="javascript">$sample1 /data/cDNA1_R1.fq.gz;/data/cDNA1_R2.fq.gz /data/oligo1_R1.fq.gz,/data/oligo4_R1.fq.gz;/data/oligo1_R2.fq.gz,/data/oligo4_R2.fq.gz $sample2 /data/cDNA2_R1.fq.gz;/data/cDNA2_R2.fq.gz /data/oligo2_R1.fq.gz;/data/oligo2_R2.fq.gz $sample3 /data/cDNA3_R1.fq.gz;/data/cDNA3_R2.fq.gz /data/oligo3_R1.fq.gz;/data/oligo3_R2.fq.gz</code>
我们现在需要根据样本文件名规律来生成示例文件的对应信息。手写是不可能手写的,容易出错不说,还不能重复。这里通常需要我们来编程批量进行文本处理。
DeepSeek 助力初步观察这个需求实现起来还是挺复杂的,以前往往需要花费一定时间来进行代码实现。现在AI盛行,这个时候我们就可以使用DeepSeek来快速生成我们需要的代码。比如下面的代码:
代码语言:javascript代码运行次数:0运行复制<code class="javascript">ls *.gz | awk -F_ 'BEGIN {OFS="\t"} { sample = $1; lib = $2; split($3, temp, "."); direction = temp[1]; all_samples[sample] = 1; if (lib ~ /^L[0-9]+/) { lib_num = substr(lib, 2) + 0; # 提取L后面的数字 # 记录文库顺序 if (!(sample SUBSEP lib_num in lib_seen)) { libs_order[sample] = libs_order[sample] ? libs_order[sample] " " lib_num : lib_num; lib_seen[sample, lib_num] = 1; } # 存储文件路径 if (direction == "f1") { f1_files[sample, lib_num] = $0; } else if (direction == "r2") { r2_files[sample, lib_num] = $0; } } else if (lib == "oligo") { if (direction == "f1") { oligo_f1[sample] = $0; } else if (direction == "r2") { oligo_r2[sample] = $0; } }}END { for (sample in all_samples) { # 处理L00数据 l00_f1 = ""; l00_r2 = ""; if (sample in libs_order) { split(libs_order[sample], lib_nums, " "); n = length(lib_nums); # 冒号排序确保文库顺序 for (i=1; i<=n; i++) { for (j=1; j<=n-i; j++) { if (lib_nums[j+1] < lib_nums[j]) { tmp = lib_nums[j]; lib_nums[j] = lib_nums[j+1]; lib_nums[j+1] = tmp; } } } # 构建f1和r2列表 for (i=1; i<=n; i++) { num = lib_nums[i]; if ((sample, num) in f1_files) { l00_f1 = l00_f1 ? l00_f1 "," f1_files[sample, num] : f1_files[sample, num]; } if ((sample, num) in r2_files) { l00_r2 = l00_r2 ? l00_r2 "," r2_files[sample, num] : r2_files[sample, num]; } } } # 合并L00字段 l00_combined = l00_f1 ";" l00_r2; # 处理Oligo数据 oligo_data = ""; if (oligo_f1[sample] && oligo_r2[sample]) { oligo_data = oligo_f1[sample] ";" oligo_r2[sample]; } print sample, l00_combined, oligo_data; }}' > sample2.tsv</code>快速得到我们需要的样本对应信息文件sample2.tsv :(检查文件信息)
<code class="javascript">$cat sample2.tsv D10 D10_L001_f1.fq.gz,D10_L002_f1.fq.gz;D10_L001_r2.fq.gz,D10_L002_r2.fq.gz D10_oligo_f1.fq.gz;D10_oligo_r2.fq.gzD15 D15_L001_f1.fq.gz,D15_L002_f1.fq.gz;D15_L001_r2.fq.gz,D15_L002_r2.fq.gz D15_oligo_f1.fq.gz;D15_oligo_r2.fq.gzD5-2 D5-2_L001_f1.fq.gz,D5-2_L002_f1.fq.gz;D5-2_L001_r2.fq.gz,D5-2_L002_r2.fq.gz D5-2_oligo_f1.fq.gz;D5-2_oligo_r2.fq.gzD2-1 D2-1_L001_f1.fq.gz,D2-1_L002_f1.fq.gz;D2-1_L001_r2.fq.gz,D2-1_L002_r2.fq.gz D2-1_oligo_f1.fq.gz;D2-1_oligo_r2.fq.gzD8-2 D8-2_L001_f1.fq.gz,D8-2_L002_f1.fq.gz;D8-2_L001_r2.fq.gz,D8-2_L002_r2.fq.gz D8-2_oligo_f1.fq.gz;D8-2_oligo_r2.fq.gzD5-1 D5-1_L001_f1.fq.gz,D5-1_L002_f1.fq.gz;D5-1_L001_r2.fq.gz,D5-1_L002_r2.fq.gz D5-1_oligo_f1.fq.gz;D5-1_oligo_r2.fq.gzD2-2 D2-2_L001_f1.fq.gz,D2-2_L002_f1.fq.gz;D2-2_L001_r2.fq.gz,D2-2_L002_r2.fq.gz D2-2_oligo_f1.fq.gz;D2-2_oligo_r2.fq.gzD12 D12_L001_f1.fq.gz;D12_L001_r2.fq.gz D12_oligo_f1.fq.gz;D12_oligo_r2.fq.gzD8-1 D8-1_L001_f1.fq.gz,D8-1_L002_f1.fq.gz;D8-1_L001_r2.fq.gz,D8-1_L002_r2.fq.gz D8-1_oligo_f1.fq.gz;D8-1_oligo_r2.fq.gz</code>
然后就是批量生成运行脚本代码语言:javascript代码运行次数:0运行复制
<code class="javascript">dnbc4tools rna multi --list sample2.tsv --genomeDir ~/reference/human/homo_ensembl_112_dnbc4_index --threads 10</code>
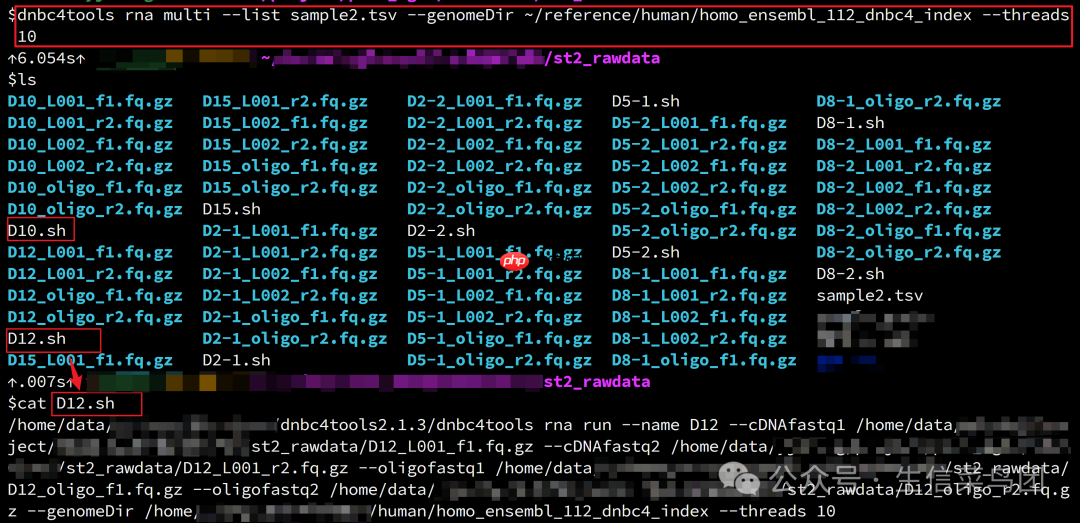 示例
示例示例
至此,后面提交批量运行任务即可。详见:
DNBC4tools—华大DNBelab系列单细胞分析pipeline玩转服务器—从前台到后台,让你的任务无忧运行以上就是DeepSeek助力快速生成DNBC4tools所需样本对应信息的详细内容,更多请关注php中文网其它相关文章!

Copyright 2014-2025 https://www.php.cn/ All Rights Reserved | php.cn | 湘ICP备2023035733号