☞☞☞AI 智能聊天, 问答助手, AI 智能搜索, 免费无限量使用 DeepSeek R1 模型☜☜☜

1. 引言
空间组学已扩展了分子类别分析的范围,但许多技术受到空间分辨率的限制。现有计算方法主要针对转录组学数据,缺乏对新兴空间组学技术的适应性。
2. soScope 框架
北京航空航天大学和清华大学的研究人员提出了 soScope,这是一个统一的生成框架,旨在提高空间组学数据的质量和分辨率。
3. 技术原理
soScope 汇总来自组学、空间关系和图像的多模态组织信息。通过分布先验与组学特定建模的联合推断,输出具有增强分辨率的组学谱。
4. 性能评估
soScope 在 Visium、Xenium、spatial-CUT&Tag、slide-DNA/RNA-seq 等平台上的评估结果表明:
5. 扩展应用
soScope 已扩展到 spatial-CITE-seq 和空间 ATAC-RNA-seq,利用跨组学参考进行多组学增强。
6. 结论
soScope 提供了一种多功能工具,提高了空间组学技术和资源的利用率。
7. 参考
该研究已于 2024 年 8 月 2 日以「Tissue characterization at an enhanced resolution across spatial omics platforms with deep generative model」为题发表在《Nature Communications》。

组织是由具有不同分子状态和空间组织的细胞构成的。空间组学技术近年来取得了显著进展,可以在保持空间背景的同时,对各种分子类别进行空间分析。
挑战和解决方案
尽管取得了早期成功,但空间组学技术仍面临两个主要挑战:
计算技术可以改善空间组学数据的分辨率,但目前的方法大多仅针对单一组织模态,难以充分利用多模态信息。
soScope:提高空间分辨率和数据质量
北航和清华的研究团队引入了空间组学范围(soScope),这是一个完全生成的框架,它模拟来自不同空间组学技术的点级概况的生成过程,旨在提高它们的空间分辨率和数据质量。
soScope将每个点视为增强空间分辨率的“子点”的集合,其组学特征与空间位置和形态模式相关。然后,soScope使用多模态深度学习框架整合斑点组学概况、空间关系和高分辨率形态学图像,并联合推断子斑点分辨率下的组学概况。通过选择组学特定分布,soScope可以对不同的空间组学数据进行精确建模和减少变异。
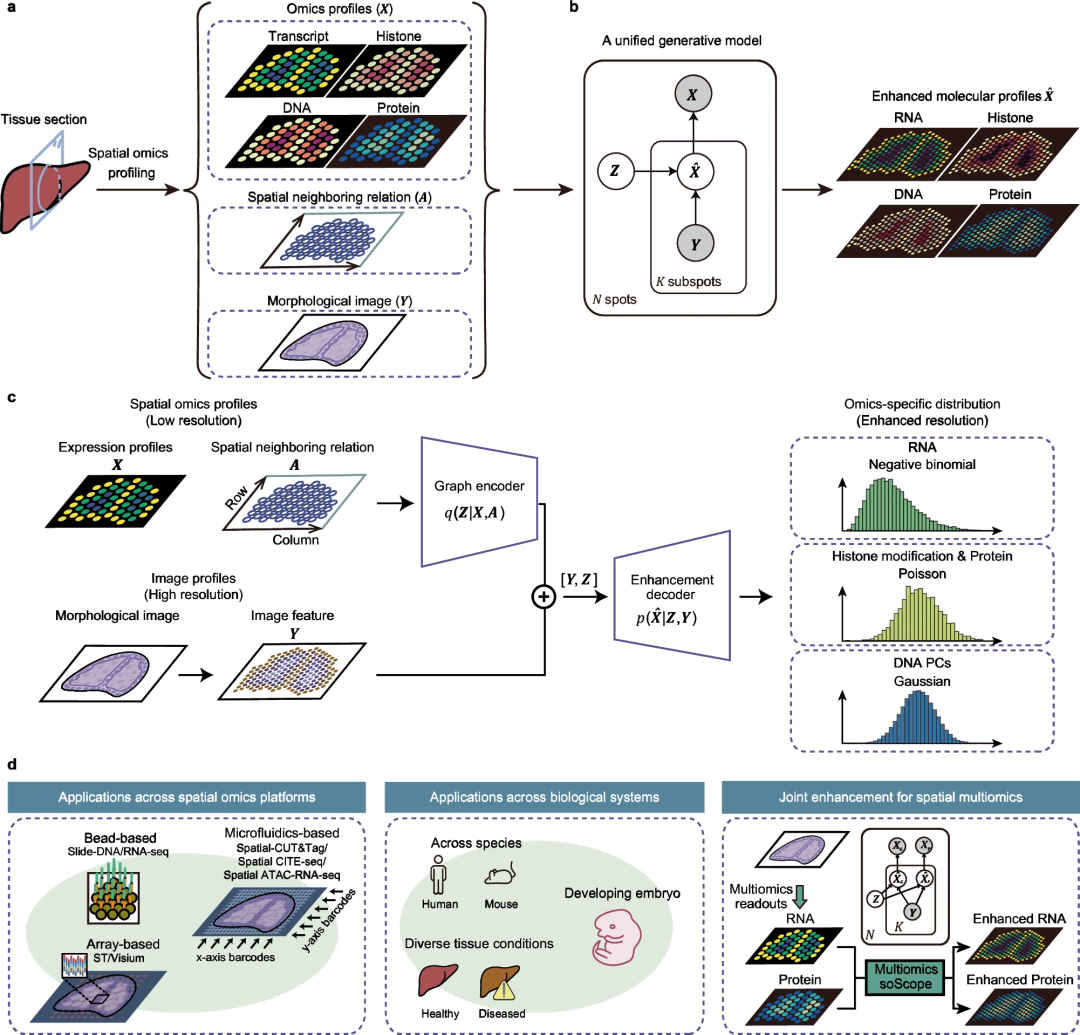
soScope 特点:
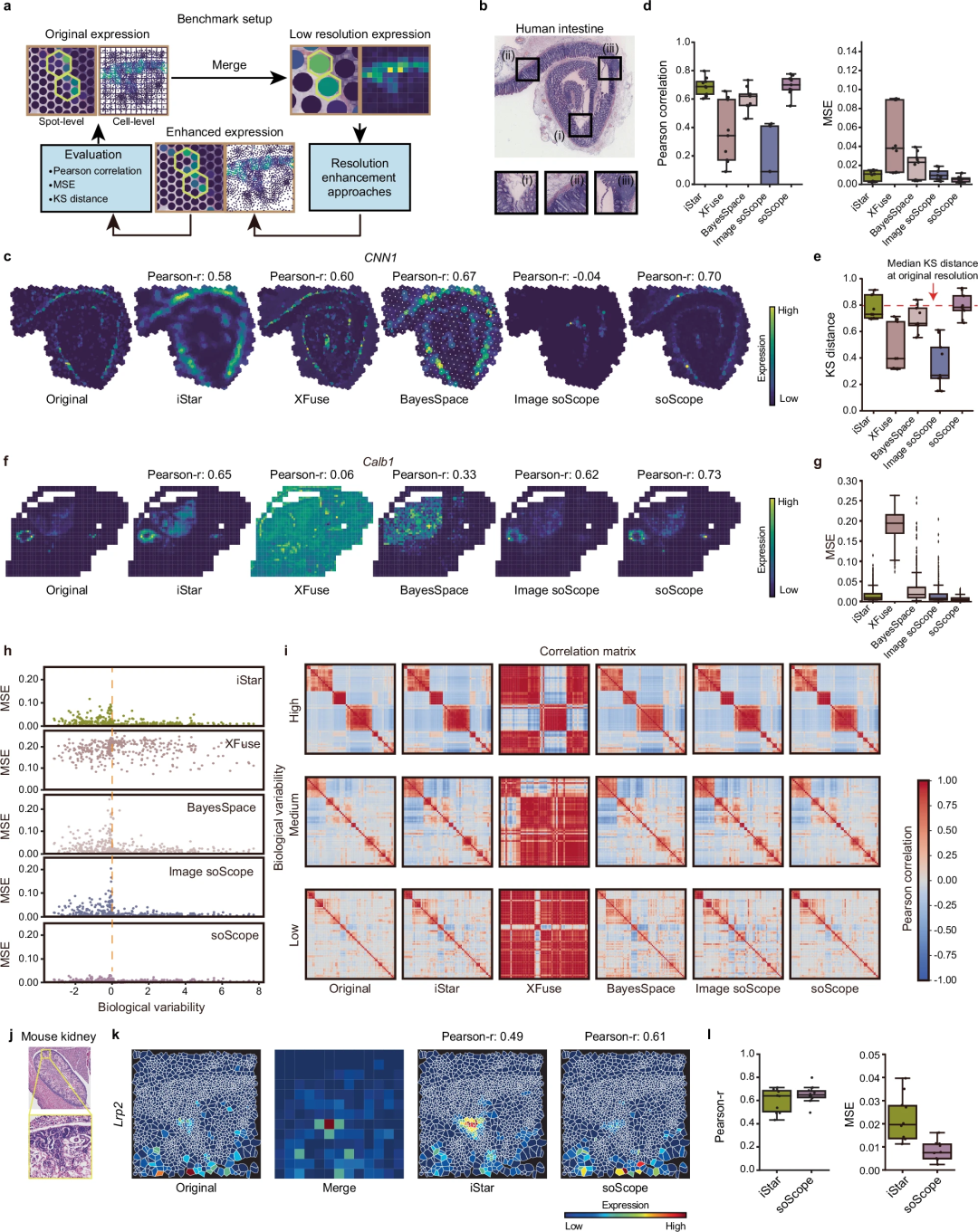
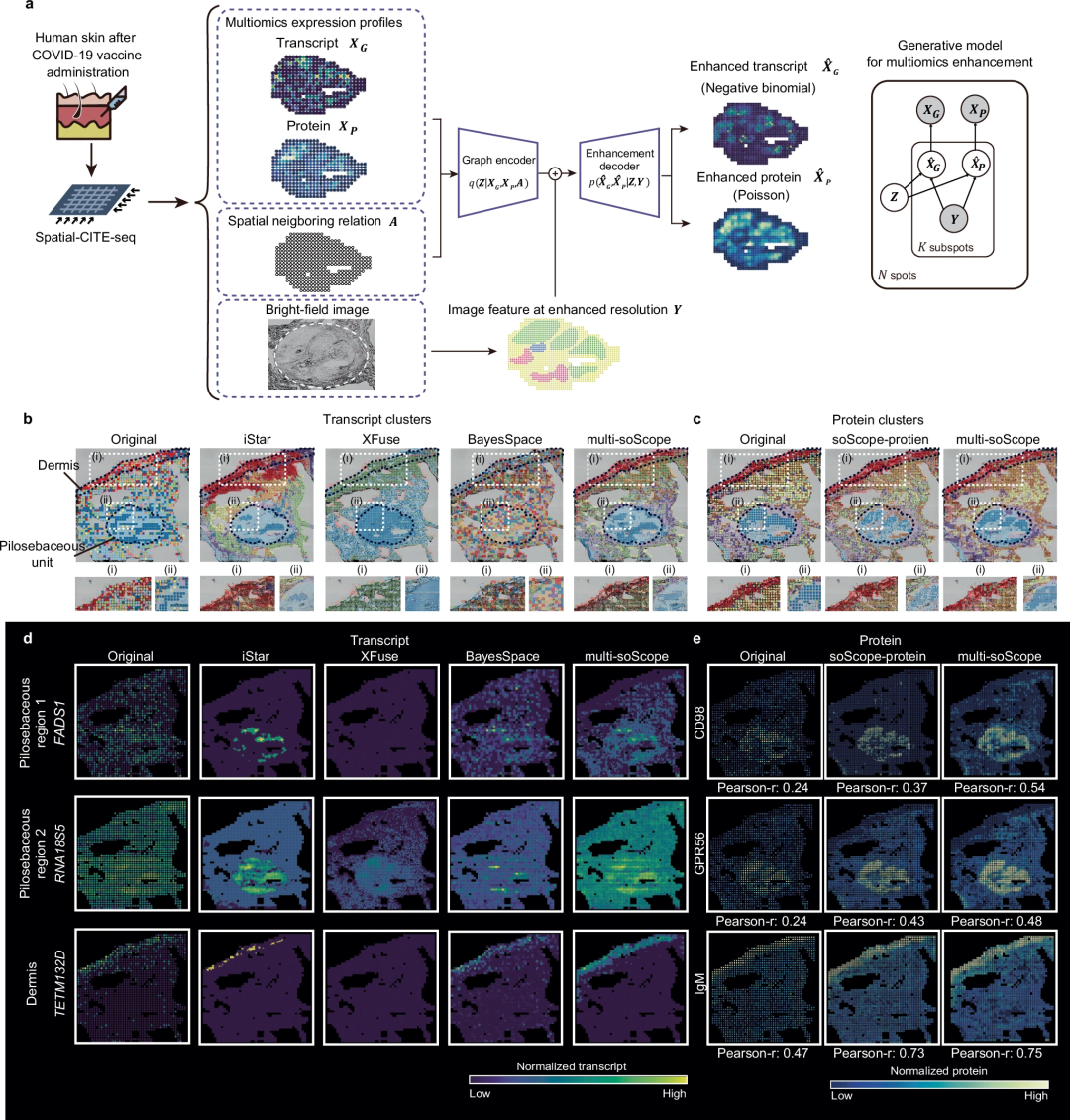
该团队广泛评估了 soScope 对通过多种空间技术分析的多种分子类型的有效性和普遍性,包括 Visium、Xenium、spatial-CUT&Tag、slide-DNA-seq、slide-RNA-seq、spatial-CITE-seq 和 spatial ATAC-RNA-seq。
在健康和患病组织中,soScope 改进了组织域识别,提高了已知标记的可区分性,并纠正了数据和技术偏差。该方法能够揭示比原始分辨率高出 36 倍的更精细的组织结构。它可以有效地适应空间多组学数据,以同时增强多组学概况。
研究人员注意到,有几种基于成像的空间组学技术,例如 seqFISH、STARmap 和 MERFISH,它们可以直接实现单细胞分辨率的空间分析,但代价是组学通量较低和组织区域较小。虽然 soScope 为预先指定的亚点或细胞位置提供了增强的配置文件,但它可能无法达到亚细胞分辨率。
进一步提高分辨率:
降低计算成本:
对于包含来自同一器官的多个连续切片的较大数据集,soScope 可以:
潜力:
随着空间组学数据资源的不断扩展和新空间技术的出现,研究人员相信 soScope 具有以下潜力:
论文链接:
https://www.nature.com/articles/s41467-024-50837-5
以上就是比原始分辨率高36倍,北航、清华团队用AI在多空间组学平台上高分辨率表征组织,登Nature子刊的详细内容,更多请关注php中文网其它相关文章!

每个人都需要一台速度更快、更稳定的 PC。随着时间的推移,垃圾文件、旧注册表数据和不必要的后台进程会占用资源并降低性能。幸运的是,许多工具可以让 Windows 保持平稳运行。




Copyright 2014-2025 https://www.php.cn/ All Rights Reserved | php.cn | 湘ICP备2023035733号