前言
 设计的初衷在目前的细胞类型鉴定工具中,支持向量机(SVM)的准确性超过了大多数监督注释方法。然而,由于监督注释方法在大多数单细胞数据中缺乏真实参照,因此其易用性不如非监督方法,这也是非监督方法占主流的原因之一。使用非监督方法时,需要人工介入,调整分群的分辨率,并提供标记基因,这会导致选择标记基因耗时且重复性差(因为不同的人选择的标记基因可能不同)。
设计的初衷在目前的细胞类型鉴定工具中,支持向量机(SVM)的准确性超过了大多数监督注释方法。然而,由于监督注释方法在大多数单细胞数据中缺乏真实参照,因此其易用性不如非监督方法,这也是非监督方法占主流的原因之一。使用非监督方法时,需要人工介入,调整分群的分辨率,并提供标记基因,这会导致选择标记基因耗时且重复性差(因为不同的人选择的标记基因可能不同)。
为了解决第一个问题(选择标记基因耗时),有人建立了专门收录这些基因的数据库,如PanglaoDB和CellMarker,涵盖了人和小鼠多种细胞类型的标记基因;此外,NeuroExpresso是大脑组织的标记基因数据库。
这个工具MACA,全称为基于标记的自动细胞类型注释工具,旨在提高细胞注释的速度和准确性。
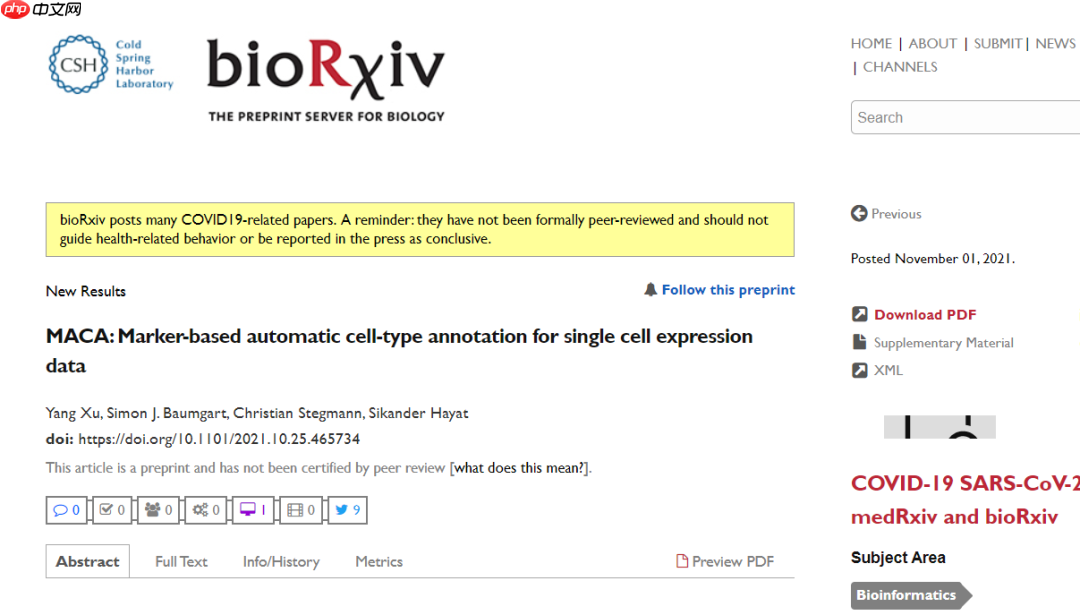
设计的结构MACA的设计结构如下:首先,读取单细胞或单核RNA-Seq的表达矩阵。
然后,为每个细胞计算两个标签:
首先,结合标记数据库,使用原始计数表达矩阵,计算每个细胞的细胞类型分数 =》 将基因表达矩阵转换为细胞类型分数矩阵。然后,对每个细胞分配细胞类型:将分数最高的细胞类型分配给该细胞 =》 产生标签1。同时,利用细胞类型分数矩阵和Louvain社区检测算法,将细胞聚类 =》 产生标签2(即某群细胞属于什么类型)。由于一开始不知道具体有多少种细胞类型,MACA默认将分辨率调高,以避免许多同源细胞被分成许多小的聚类。
随后,通过一系列统计知识,利用标签1和标签2:MACA记录每个聚类中基于细胞类型分数的显著或至少前三的细胞类型。
意思是挑选出个体表现突出且整体符合要求的那组细胞。
 实际测试使用的方法、数据研究了4种已提出的将基因表达矩阵转换为细胞类型分数矩阵的评分方法,使用了2个公共标记数据库,分析了6个单细胞研究(包含3000至20000个细胞)。使用调整后的兰德指数(ARI)和归一化互信息(NMI)进行评测。评测结果显示,在所有6个数据集中,使用PanglaoDB中的标记和PlinerScore的注释一致性最高。因此,采用PlinerScore作为评分方法。
实际测试使用的方法、数据研究了4种已提出的将基因表达矩阵转换为细胞类型分数矩阵的评分方法,使用了2个公共标记数据库,分析了6个单细胞研究(包含3000至20000个细胞)。使用调整后的兰德指数(ARI)和归一化互信息(NMI)进行评测。评测结果显示,在所有6个数据集中,使用PanglaoDB中的标记和PlinerScore的注释一致性最高。因此,采用PlinerScore作为评分方法。
接下来,与自动细胞注释工具进行比较,使用PanglaoDB的标记,与CellAssign、SCINA、Cell-ID和scCATCH进行比较。
发现速度差异:
MACA可以在1分钟内完成3000个细胞的注释,对于相对较大的数据集(最多20000个细胞),不到2分钟即可完成。scCATCH和Cell-ID的耗时比MACA长,SCINA在大数据集上大约需要20分钟,而CellAssign在超过20000个细胞时由于内存不足耗时最长。
发现注释结果差异:
MACA标注的细胞与CellAssign、SCINA、Cell-ID和scCATCH相比,具有更高的共识度。与作者的结果相似:MACA和scCATCH识别出的细胞类型数量与作者的注释相似。






























