在处理fasta序列时,我们常常需要知道每条序列的长度。今天小编将与大家分享四种获取fasta序列长度的方法。
一、使用awk
以下是使用awk获取fasta序列长度的代码:
awk '/^>/{if (l!="") print l; print; l=0; next}{l+=length($0)}END{print l}' test.fasta该命令的输出如下:
>Chr115704606>Chr108327059>Chr118696443>Chr127853492>Chr134340075>Chr146288713>Chr29509017>Chr311222275>Chr47647452>Chr57499939>Chr64872821>Chr78973615>Chr88275968>Chr98318069
二、使用bioawk
安装bioawk并使用以下命令获取fasta序列长度:
conda install bioawkbioawk -c fastx '{ print $name, length($seq) }' 输出结果如下:
Chr1 15704606Chr10 8327059Chr11 8696443Chr12 7853492Chr13 4340075Chr14 6288713Chr2 9509017Chr3 11222275Chr4 7647452Chr5 7499939Chr6 4872821Chr7 8973615Chr8 8275968Chr9 8318069
三、使用samtools
首先生成.fai文件,然后提取前两列:
#生成.fai文件samtools faidx test.fasta#提取前两列cut -f1-2 test.fasta.fai
生成的.fai文件如下,前两列显示fasta序列的名字和长度。
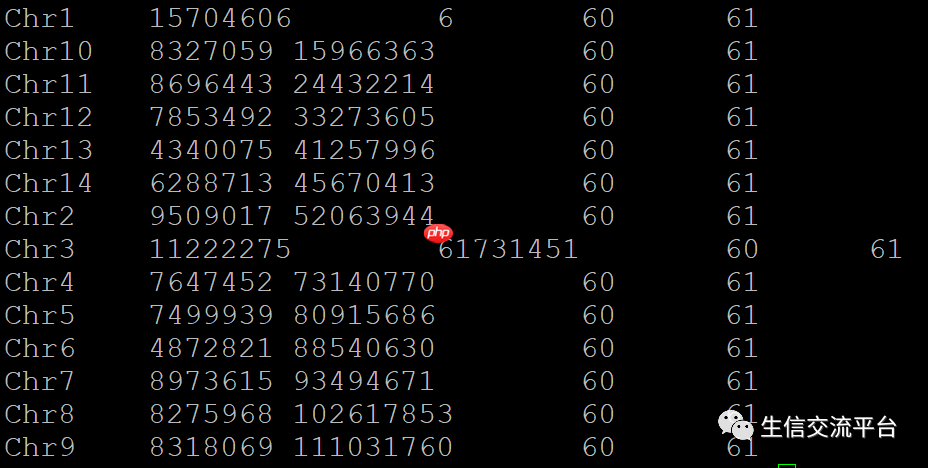
.fai文件的每一列具体含义:
第一列 NAME : 序列的名称,只保留“>”后,第一个空白之前的内容;第二列 LENGTH: 序列的长度, 单位为bp;第三列 OFFSET : 第一个碱基的偏移量, 从0开始计数,换行符也统计进行;第四列 LINEBASES : 除了最后一行外, 其他代表序列的行的碱基数, 单位为bp;第五列 LINEWIDTH : 行宽, 除了最后一行外, 其他代表序列的行的长度, 包括换行符,
四、使用seqkit
安装seqkit并使用以下命令获取fasta序列长度:
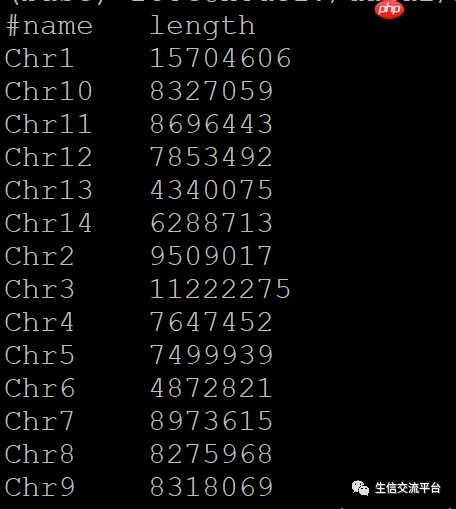
conda install seqkitseqkit fx2tab --length --name --header-line test.fasta
以上就是四种获取fasta序列长度的方法的详细内容,更多请关注php中文网其它相关文章!

每个人都需要一台速度更快、更稳定的 PC。随着时间的推移,垃圾文件、旧注册表数据和不必要的后台进程会占用资源并降低性能。幸运的是,许多工具可以让 Windows 保持平稳运行。

Copyright 2014-2025 https://www.php.cn/ All Rights Reserved | php.cn | 湘ICP备2023035733号