本文介绍了一个基于飞桨框架的核酸统计项目。受复旦博士用代码核查核酸报告启发,该项目在参考项目基础上修改错误、压缩行数、优化排版,仅用7行代码实现核酸图像统计并生成CSV结果。包含库函数安装与功能实现,将JPG截图上传至指定文件夹一键运行即可生成统计表格。
☞☞☞AI 智能聊天, 问答助手, AI 智能搜索, 免费无限量使用 DeepSeek R1 模型☜☜☜

7行代码带你快速实现核酸统计表格生成
使用该项目展示PADDLE框架的便捷易用。
近日,人民网等主流媒体广泛报道了新闻《复旦博士用130行代码核查核酸报告,800份只需要2分钟》。
报道中,复旦大学信息科学与工程学院生物医学工程专业博士生仅使用130代码就完成了数百人的核酸截图核查工作,引发全网的热议。
随着该新闻迅速出圈,借助飞桨的预训练模型,也有不断的作者试图用更少的代码来实现这一功能。
从50行到10行,在保证功能的情况下,代码越来越精简。
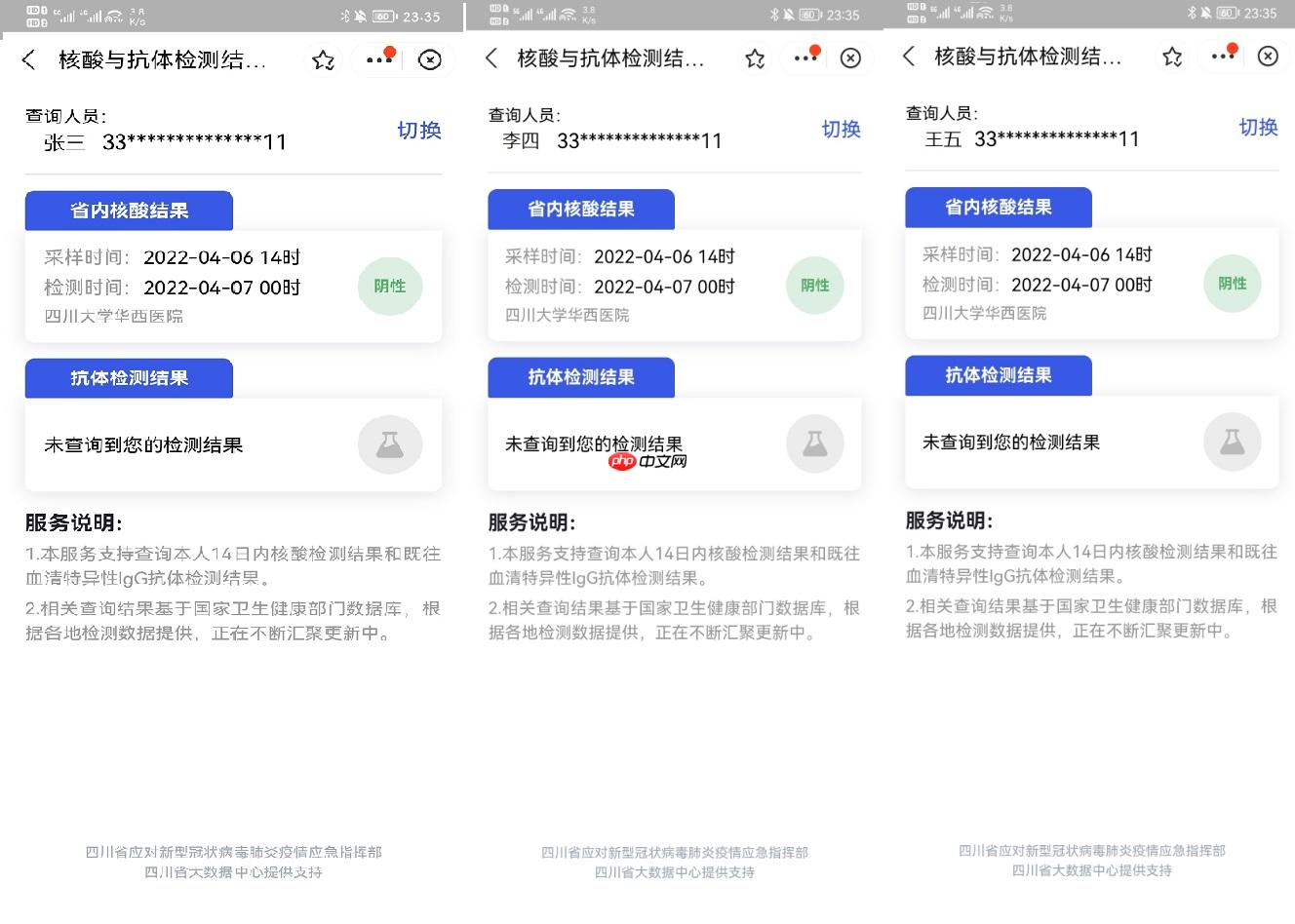
笔者在前人的基础上,进一步精简代码,完成该项目。对如下的核酸图像进行统计生成CSV统计结果
项目主要工作:
修改参考项目的编码错误
压缩参考项目代码行数
优化参考项目排版
参考项目
项目包括所用库函数的安装以及功能实现
飞桨仅使用七行代码就能实现核酸检测统计结果生成
!pip uninstall paddlehub -y !pip install paddlehub shapely pyclipper %env CUDA_VISIBLE_DEVICES=0%matplotlib inline
import paddlehub as hub ,cv2,os,csv,glob
results = hub.Module(name="chinese_ocr_db_crnn_server").recognize_text(images=[cv2.imread(image_path) for image_path in glob.glob("images/*.jpg")] , use_gpu=True, output_dir='ocr_result', visualization=True, box_thresh=0.5, text_thresh=0.5)with open('result.csv', 'w',encoding='utf-8-sig') as f:
f.write('姓名_身份证号码,采样时间,检测时间,结果\n') for result in results:
data = [str(i['text'])+"," if(("**" in i['text']) or ("时间" in i['text']) or ("阴性" in i['text'])) else '' for i in result['data']]
f.write(''.join(data)+"\n")[2022-04-16 23:55:54,416] [ WARNING] - The _initialize method in HubModule will soon be deprecated, you can use the __init__() to handle the initialization of the object [2022-04-16 23:55:55,105] [ WARNING] - The _initialize method in HubModule will soon be deprecated, you can use the __init__() to handle the initialization of the object
该部分包括核酸统计表的生成的效果和项目使用说明
生成效果
我们展示核酸截图和生成效果对比:
| 示例数据 | CSV效果 |
|---|---|
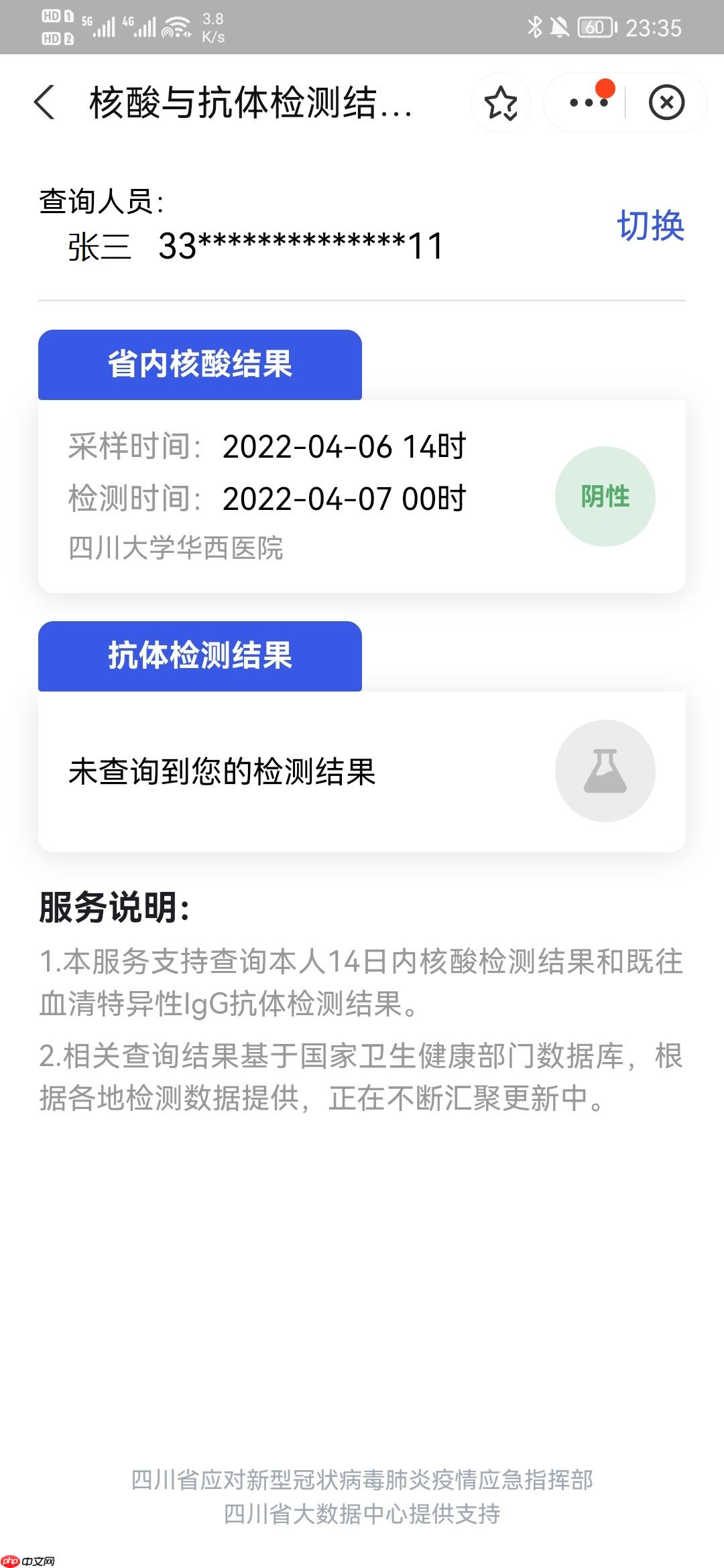
|
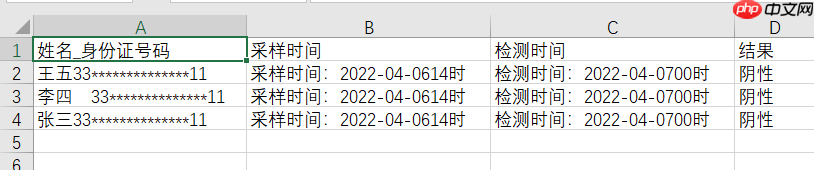
|
将JPG格式截图上传至images文件夹下一键运行,稍等,result.csv即为统计表格。
以上就是强无敌!飞桨仅7行完成社群日常核酸统计的详细内容,更多请关注php中文网其它相关文章!

每个人都需要一台速度更快、更稳定的 PC。随着时间的推移,垃圾文件、旧注册表数据和不必要的后台进程会占用资源并降低性能。幸运的是,许多工具可以让 Windows 保持平稳运行。

 C++高性能并发应用_C++如何开发性能关键应用
C++高性能并发应用_C++如何开发性能关键应用
 Java AI集成Deep Java Library_Java怎么集成AI模型部署
Java AI集成Deep Java Library_Java怎么集成AI模型部署
 Golang后端API开发_Golang如何高效开发后端和API
Golang后端API开发_Golang如何高效开发后端和API
 Python异步并发改进_Python异步编程有哪些新改进
Python异步并发改进_Python异步编程有哪些新改进
 C++系统编程内存管理_C++系统编程怎么与Rust竞争内存安全
C++系统编程内存管理_C++系统编程怎么与Rust竞争内存安全
 Java GraalVM原生镜像构建_Java怎么用GraalVM构建高效原生镜像
Java GraalVM原生镜像构建_Java怎么用GraalVM构建高效原生镜像
 Python FastAPI异步API开发_Python怎么用FastAPI构建异步API
Python FastAPI异步API开发_Python怎么用FastAPI构建异步API
 C++现代C++20/23/26特性_现代C++有哪些新标准特性如modules和coroutines
C++现代C++20/23/26特性_现代C++有哪些新标准特性如modules和coroutines
Copyright 2014-2025 https://www.php.cn/ All Rights Reserved | php.cn | 湘ICP备2023035733号