电子病历、人工智能、物联网设备、5G技术的快速演化,与大规模的数据资源,成为了数字化医疗的全新支撑。 对于医药公司、医疗机构、科技公司,如何整合全新的技术能力、人才资源、数据资源,为数字化医疗的创新提供更强的动力,成为了共同关切的重要话题。
☞☞☞AI 智能聊天, 问答助手, AI 智能搜索, 免费无限量使用 DeepSeek R1 模型☜☜☜

食品与疾病关系预测算法赛道:baseline
一、赛题背景
本次数字医疗算法应用创新大赛是对于这一重要话题的主动探索,旨在最大化利用当前高速发展的算法技术与数据资源,培养数字化医疗人才、激励数字化医疗创新。大赛双赛道分别为食品与疾病关系预测算法赛道和生物共融与数字疗法应用赛道。
1.1 赛题任务
本赛道将提供脱敏后的食物与疾病特征,参赛团队根据主办方提供数据,在高度稀疏数据的场景中,进一步挖掘、融合特征并设计模型,以预测食物与疾病的关系。初赛阶段为二分类问题,分类标签分别为 0(无关)、1(存在正面或负面的影响)。
1.2 赛题数据简介
本次算法赛将提供超过 23.5W 的食物、疾病对应关系及其量化得分,其中食物特征超过 200 个,疾病特征由 3 种不同的方式抽取,累积超过 4000 个特征信息。初赛为 0、1 二分类预测,提供食物、疾病特征,与食物疾病的关系标签。
复赛阶段同时评估 0、1 二分类与相关性评级,在原训练集中增加食物疾病相关性评级的标签数据。
1.2.1 训练集
训练集包括疾病特征数据、食物特征数据(共计 348 种食物)、以及食物疾病关系,用于模型训练:
- 疾病特征集:disease_feature1.csv、disease_feature2.csv、disease_feature3.csv
- 食物特征集:train_food.csv
- 食物疾病关系:train_answer.csv
- 「复赛阶段」食物与疾病关系的相关性评级:semi_train_answer.csv
1.2.2 测试集
初赛测试集分两个阶段(A/B 榜),不提供预测结果,其中:
- 初赛第一阶段 A 榜测试集: 2023 年 2 月 22 日中午 12:00:00— 2023 年 3 月 20 日中午 12:00:00,包括 A 榜阶段食物特征数据(共计 115 种食物)与初赛 A 榜提交样例,用于模型结果验证:
preliminary_a_food.csv preliminary_a_submit_sample.csv
- 初赛第二阶段 B 榜测试集: 2023 年 3 月 20 日中午 12:00:00— 2023 年 3 月 22 日中午 12:00:00,包括 B 榜阶段食物特征数据(共计 116 种食物)与初赛 B 榜提交样例,用于模型结果验证:
preliminary_b_food.csvpreliminary_b_submit_sample.csv
- 初赛第二阶段 B 测试集与初赛第一阶段 A 榜测试集分布与规模相同,将于 B 榜提交开始后在赛事主页提供下载,最终初赛排名以初赛第二阶段 B 榜成绩为准。
1.3 字段说明
1.3.1 疾病特征
累计包含 407 种疾病的 4630 种特征信息,三种不同的特征抽取方式将疾病特征划分为三部分特征集,数据高度稀疏。
| 字段名称 | 格式 | 解释说明 | 范围/特征集1 | 范围/特征集2 | 范围/特征集3 |
|---|---|---|---|---|---|
| disease_id | 字符串 | 疾病 id | 共涉及 220 种疾病 | 共涉及 301 种疾病 | 共涉及 392 种疾病 |
| F_x | 浮点型 | 疾病特征值 | F_0 ~F_4629,字段名称不连续,共涉及 996 种疾病特征 | F_0 ~F_4629,字段名称不连续,共涉及 3181 种疾病特征 | F_1 ~F_4627,字段名称不连续,共涉及 1453 种疾病特征 |
1.3.2 食物特征
| 序列 | 字段名称 | 格式 | 解释说明 | 示例 |
|---|---|---|---|---|
| 1 | food_id | 字符串 | 食物 id | food_0 |
| 2~213 | N_x | 浮点型 | 212 种食物特征,字段名称从 N_0 ~N_211 | 0.123 |
1.3.3 食物疾病关系
| 序列 | 字段名称 | 格式 | 解释说明 | 示例 |
|---|---|---|---|---|
| 1 | food_id | 字符串 | 食物 id | food_0 |
| 2 | disease_id | 字符串 | 疾病 id | disease_0 |
| 3 | related | 整型 | 食物与疾病是否相关:0(无关)、1(存在正面或负面的影响) | 0 |
1.4 评测方法
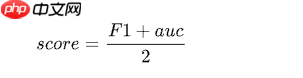
1.5 提交示例
| 序列 | 字段名称 | 格式 | 解释说明 | 示例 |
|---|---|---|---|---|
| 1 | food_id | 字符串 | 食物 id | food_0 |
| 2 | disease_id | 字符串 | 疾病 id | disease_0 |
| 3 | related_prob | 浮点型 | 食品与疾病预测为 1 的概率若 related_prob >= 0.5,评审计算 f1 得分时判定为类别 1 | 0.1 |
1.6 比赛传送门
比赛传送门
二、项目介绍
2.1 项目意义
在本次食物与疾病关系预测挑战赛中,参赛团队将获得脱敏后的大量数据集。这些数据集包含了各种类型的食物和相应的健康指标,例如血压、胆固醇等等。这些指标可以帮助我们了解不同种类的食物对人体健康产生的影响。在实际应用中,由于数据量巨大且高度稀疏,传统的特征提取方法难以有效地提取出有用信息。因此,在本次比赛中,参赛团队需要进一步挖掘、融合特征并设计模型,以预测食物与疾病的关系。
在这个 Baseline 中,我们分别尝试复杂的特征结合传统的机器学习模型。我们在解决机器学习问题时,一般会遵循以下流程: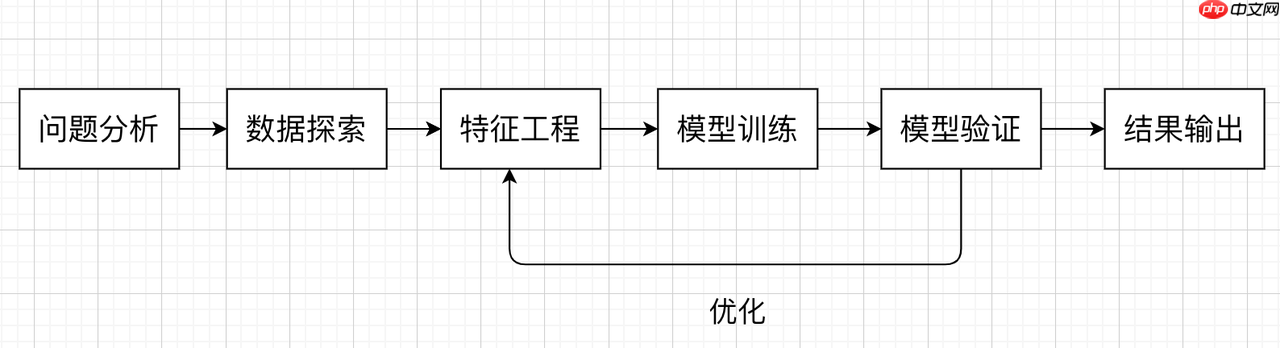
2.2 模型介绍
LightGBM(LightGBM: Gradient Boosting of Big Data Applications)是一种基于梯度提升树模型(Gradient Boosting Decision Tree)的轻量级机器学习算法,用于解决大规模数据集的分类、回归和聚类问题。它的核心思想是利用梯度下降来优化模型参数,从而提高模型的预测准确率。LightGBM的主要特点包括以下几个方面:
- 轻量级:LightGBM采用了基于梯度的优化方法,避免了过多的参数调整,从而减少了计算量和内存消耗。
- 高效性:LightGBM的基本思想是利用梯度下降来不断逼近模型的最优参数,从而减少了搜索空间,提高了模型的效率。
- 可解释性:LightGBM采用了可解释的数据预处理方法,即在训练过程中对数据进行归一化、剪枝等处理,从而提高了模型的可解释性和可靠性。
- 易用性:LightGBM提供了灵活的模型定义方式,可以根据实际需求进行定制化开发,同时也提供了丰富的可视化工具,便于用户理解和操作。
- 通用性:LightGBM可以适用于多种机器学习框架和数据存储格式,具有广泛的应用前景。
- 总之,LightGBM是一种高效、可解释、易用的轻量级机器学习算法,适用于大规模数据集的分类、回归和聚类问题。
三、详细方案实现
3.1 数据分析
3.1.1 数据解压缩
!unzip -d /home/aistudio/data/data200766/ /home/aistudio/data/data200766/初赛A榜测试集.zip!unzip -d /home/aistudio/data/data200766/ /home/aistudio/data/data200766/初赛B榜测试集.zip!unzip -d /home/aistudio/data/data200766/ /home/aistudio/data/data200766/训练集.zip
Archive: /home/aistudio/data/data200766/初赛A榜测试集.zip creating: /home/aistudio/data/data200766/初赛A榜测试集/ inflating: /home/aistudio/data/data200766/初赛A榜测试集/preliminary_a_submit_sample.csv inflating: /home/aistudio/data/data200766/初赛A榜测试集/preliminary_a_food.csv Archive: /home/aistudio/data/data200766/初赛B榜测试集.zip creating: /home/aistudio/data/data200766/初赛B榜测试集/ inflating: /home/aistudio/data/data200766/初赛B榜测试集/preliminary_b_submit_sample.csv inflating: /home/aistudio/data/data200766/初赛B榜测试集/preliminary_b_food.csv Archive: /home/aistudio/data/data200766/训练集.zip creating: /home/aistudio/data/data200766/训练集/ inflating: /home/aistudio/data/data200766/训练集/disease_feature3.csv inflating: /home/aistudio/data/data200766/训练集/disease_feature2.csv inflating: /home/aistudio/data/data200766/训练集/disease_feature1.csv inflating: /home/aistudio/data/data200766/训练集/train_answer.csv inflating: /home/aistudio/data/data200766/训练集/train_food.csv
3.1.2 导入相关库
!pip install --upgrade pip !git clone --recursive https://github.com/Microsoft/LightGBM !cd LightGBM && rm -rf build && mkdir build && cd build && cmake -DUSE_GPU=1 ../../LightGBM && make -j4 && cd ../python-package && python3 setup.py install --precompile --gpu;
import pandas as pdimport osimport gcimport lightgbm as lgbfrom sklearn.linear_model import SGDRegressor, LinearRegression, Ridgefrom sklearn.preprocessing import MinMaxScalerimport mathimport numpy as npfrom tqdm import tqdmfrom sklearn.model_selection import StratifiedKFold, KFold, GroupKFoldfrom sklearn.metrics import accuracy_score, f1_score, roc_auc_score, log_lossimport matplotlib.pyplot as pltimport timeimport warnings
warnings.filterwarnings('ignore')
3.1.3 加载数据
disease_feature1 = pd.read_csv("/home/aistudio/data/data200766/训练集/disease_feature1.csv")
disease_feature2 = pd.read_csv("/home/aistudio/data/data200766/训练集/disease_feature2.csv")
disease_feature3 = pd.read_csv("/home/aistudio/data/data200766/训练集/disease_feature3.csv")
train_answer = pd.read_csv("/home/aistudio/data/data200766/训练集/train_answer.csv")
train_food = pd.read_csv("/home/aistudio/data/data200766/训练集/train_food.csv")
preliminary_a_food = pd.read_csv("/home/aistudio/data/data200766/初赛B榜测试集/preliminary_b_food.csv")
preliminary_a_submit_sample = pd.read_csv("/home/aistudio/data/data200766/初赛B榜测试集/preliminary_b_submit_sample.csv")
pd.set_option('display.max_columns', None)
del preliminary_a_submit_sample['related_prob'] data = pd.concat([train_answer, preliminary_a_submit_sample], axis = 0).reset_index(drop=True) data.head()
food_id disease_id related 0 food_0 disease_998 0.0 1 food_0 disease_861 0.0 2 food_0 disease_559 0.0 3 food_0 disease_841 0.0 4 food_0 disease_81 0.0
这里直接使用每个变量后的数字进行编码,当然也可以使用labelencoder的方式。
data['food'] = data['food_id'].apply(lambda x : int(x.split('_')[1]))
data['disease'] = data['disease_id'].apply(lambda x : int(x.split('_')[1]))
food = pd.concat([train_food, preliminary_a_food], axis = 0).reset_index(drop=True) food.head()
food_id N_0 N_1 N_2 N_3 N_4 N_5 N_6 N_7 N_8 N_9 N_10 \
0 food_0 NaN NaN NaN NaN 0.0 NaN NaN NaN NaN NaN NaN
1 food_1 NaN NaN NaN NaN 0.0 NaN NaN NaN NaN NaN NaN
2 food_4 NaN NaN NaN NaN 0.0 NaN NaN NaN NaN NaN NaN
3 food_5 NaN NaN NaN 0.068 0.0 0.045 0.75 0.314 NaN NaN NaN
4 food_6 NaN NaN NaN 0.115 0.0 0.091 0.58 0.508 NaN NaN 0.6
N_11 N_12 N_13 N_14 N_15 N_16 N_17 N_18 N_19 N_20 N_21 N_22 \
0 NaN NaN 0.0 32.0 NaN NaN 2.10 NaN 6.0 87.0 NaN 0.0
1 NaN NaN 0.0 268.0 NaN NaN 21.01 NaN 0.0 1.0 NaN 0.0
2 NaN NaN 0.0 62.0 NaN NaN 79.32 NaN 0.0 0.0 NaN 0.0
3 NaN NaN 0.0 13.0 NaN NaN 11.12 NaN 19.0 1094.0 NaN 0.0
4 NaN NaN 0.0 24.0 NaN NaN 3.88 NaN 9.0 449.0 NaN 0.0
N_23 N_24 N_25 N_26 N_27 N_28 N_29 N_30 N_31 N_32 N_33 N_34 \
0 NaN NaN NaN NaN NaN 14.4 NaN NaN NaN NaN 0.157 NaN
1 NaN NaN NaN NaN NaN 52.1 NaN NaN NaN NaN 1.099 NaN
2 NaN NaN NaN NaN NaN 11.1 NaN NaN NaN NaN 0.272 NaN
3 NaN NaN NaN NaN NaN 2.8 NaN NaN NaN NaN 0.078 NaN
4 NaN NaN NaN NaN NaN 16.0 NaN NaN NaN NaN 0.189 NaN
N_35 N_36 N_37 N_38 N_39 N_40 N_41 N_42 N_43 N_44 N_45 N_46 \
0 6.0 NaN NaN NaN NaN NaN NaN 23.0 NaN NaN NaN NaN
1 0.0 NaN NaN NaN NaN NaN NaN 598.0 NaN NaN NaN NaN
2 0.0 NaN NaN NaN NaN NaN NaN 299.0 NaN NaN NaN NaN
3 104.0 NaN 0.003 NaN NaN NaN NaN 124124.5 NaN NaN NaN NaN
4 0.0 NaN 0.031 NaN NaN NaN NaN 52.5 NaN NaN NaN NaN
N_47 N_48 N_49 N_50 N_51 N_52 N_53 N_54 N_55 N_56 N_57 \
0 NaN 0.056 0.409 0.069 NaN NaN NaN NaN NaN NaN 1.9
1 NaN 33.076 12.955 4.092 NaN NaN NaN NaN NaN NaN 10.9
2 NaN 0.024 0.053 0.094 NaN NaN NaN NaN NaN NaN 4.5
3 NaN 0.170 0.077 0.027 0.0 NaN NaN NaN NaN NaN 2.0
4 NaN 0.000 0.050 0.040 0.0 NaN NaN NaN NaN NaN 2.1
N_58 N_59 N_60 N_61 N_62 N_63 N_64 N_65 N_66 N_67 N_68 N_69 \
0 NaN 36.0 36.0 36.0 0.0 NaN NaN NaN NaN NaN NaN NaN
1 NaN 55.0 55.0 55.0 0.0 NaN NaN NaN NaN NaN NaN NaN
2 NaN 5.0 5.0 5.0 0.0 NaN NaN NaN NaN NaN NaN NaN
3 NaN 9.0 9.0 9.0 0.0 0.94 0.0 NaN NaN 2.37 0.157 0.040
4 NaN 52.0 52.0 52.0 0.0 1.00 0.0 NaN NaN 0.65 0.233 0.093
N_70 N_71 N_72 N_73 N_74 N_75 N_76 N_77 N_78 N_79 N_80 \
0 NaN NaN NaN NaN 0.96 NaN NaN NaN NaN 0.0 0.0
1 NaN NaN NaN NaN 3.73 NaN NaN NaN NaN 1.0 0.0
2 NaN NaN NaN NaN 1.79 NaN NaN NaN NaN 0.0 0.0
3 NaN 0.027 NaN NaN 0.39 0.041 0.0 0.077 NaN 89.0 0.0
4 NaN 0.049 NaN NaN 2.14 0.075 0.0 0.128 NaN 710.0 0.0
N_81 N_82 N_83 N_84 N_85 N_86 N_87 N_88 N_89 N_90 N_91 \
0 NaN 27.0 NaN NaN NaN NaN NaN NaN NaN 0.000 NaN
1 NaN 279.0 NaN NaN NaN NaN NaN NaN NaN 0.259 NaN
2 NaN 36.0 NaN NaN NaN NaN NaN NaN NaN 0.001 NaN
3 0.097 10.0 NaN 0.06 0.077 0.006 NaN NaN NaN 0.000 NaN
4 0.104 14.0 NaN 0.00 0.158 0.031 0.0 NaN 0.0 0.000 NaN
N_92 N_93 N_94 N_95 N_96 N_97 N_98 N_99 N_100 N_101 N_102 \
0 NaN NaN 0.056 NaN 0.000 NaN 0.0 NaN NaN 0.481 NaN
1 NaN NaN 32.754 NaN 0.007 NaN 0.0 NaN NaN 3.637 NaN
2 NaN NaN 0.023 NaN 0.000 NaN 0.0 NaN NaN 0.766 NaN
3 NaN NaN 0.170 NaN 0.000 NaN 0.0 NaN NaN 0.600 NaN
4 0.0 NaN 0.000 NaN 0.000 NaN 0.0 NaN NaN 0.978 NaN
N_103 N_104 N_105 N_106 N_107 N_108 N_109 N_110 N_111 N_112 \
0 NaN NaN NaN 70.0 NaN NaN NaN NaN 79.0 NaN
1 NaN NaN NaN 471.0 NaN NaN NaN NaN 713.0 NaN
2 NaN NaN NaN 98.0 NaN NaN NaN NaN 744.0 NaN
3 NaN 0.240 0.052 23.0 NaN NaN 18.0 NaN 259.0 0.101
4 NaN 0.274 0.075 52.0 NaN NaN 24.0 NaN 202.0 0.071
N_113 N_114 N_115 N_116 N_117 N_118 N_119 N_120 N_121 N_122 \
0 3.99 0.234 NaN NaN NaN NaN 0.175 NaN NaN NaN
1 20.96 12.945 NaN NaN NaN NaN 0.010 NaN NaN NaN
2 3.30 0.039 NaN NaN NaN NaN 0.014 NaN NaN NaN
3 1.40 0.077 NaN NaN NaN NaN 0.000 NaN NaN NaN
4 2.20 0.040 NaN NaN NaN NaN 0.010 NaN NaN NaN
N_123 N_124124 N_125 N_126 N_127 N_128 N_129 N_130 N_131 N_132 \
0 NaN 0.0 NaN NaN NaN NaN NaN NaN 0.0 NaN
1 NaN 0.0 NaN NaN NaN NaN NaN NaN 0.0 NaN
2 NaN 0.0 NaN NaN NaN NaN NaN NaN 0.0 NaN
3 NaN 0.0 NaN NaN NaN NaN NaN NaN 0.0 NaN
4 NaN 0.0 NaN 0.0 0.0 NaN NaN NaN 0.0 NaN
N_133 N_134 N_135 N_136 N_137 N_138 N_139 N_140 N_141 N_142 \
0 NaN 0.0 NaN NaN NaN NaN NaN 0.0 NaN 0.0
1 NaN 0.0 NaN NaN NaN NaN NaN 0.0 NaN 0.0
2 NaN 0.0 NaN NaN NaN NaN NaN 0.0 NaN 0.0
3 NaN 0.0 NaN NaN NaN NaN NaN 0.0 NaN 0.0
4 NaN 0.0 NaN NaN NaN NaN NaN 0.0 NaN 0.0
N_143 N_144 N_145 N_146 N_147 N_148 N_149 N_150 N_151 N_152 \
0 NaN NaN 0.0 0.126 0.6 NaN 0.0 NaN 0.000 NaN
1 NaN NaN 0.0 1.197 2.0 NaN 0.0 NaN 0.000 NaN
2 NaN NaN 0.0 0.125 0.6 NaN 0.0 NaN 0.001 NaN
3 NaN NaN 0.0 0.040 0.1 0.083 0.0 NaN 0.000 NaN
4 NaN NaN 0.0 0.141 2.3 0.106 0.0 NaN 0.000 NaN
N_153 N_154 N_155 N_156 N_157 N_158 N_159 N_160 N_161 N_162 \
0 0.002 NaN 0.059 NaN 0.008 NaN NaN NaN NaN 0.000
1 0.019 NaN 3.348 NaN 0.704 NaN NaN NaN NaN 0.000
2 0.004 NaN 0.056 NaN 0.013 NaN NaN NaN NaN 0.007
3 0.000 NaN 0.024 NaN 0.003 NaN NaN NaN NaN 0.000
4 0.000 0.0 0.040 0.0 0.000 0.0 NaN 0.0 0.0 0.000
N_163 N_164 N_165 N_166 N_167 N_168 N_169 N_170 N_171 N_172 \
0 0.0 0.000 6.0 NaN NaN NaN 0.20 NaN NaN NaN
1 0.0 0.000 3.0 NaN NaN NaN 4.86 NaN NaN NaN
2 0.0 0.001 26.0 NaN NaN NaN 65.18 NaN NaN NaN
3 0.0 0.000 1.0 NaN NaN 5.87 9.24 NaN NaN NaN
4 0.0 0.000 2.0 NaN NaN 0.23 1.88 NaN NaN NaN
N_173 N_174 N_175 N_176 N_177 N_178 N_179 N_180 N_181 N_182 \
0 NaN NaN NaN 0.0 0.076 NaN NaN NaN NaN NaN
1 NaN NaN NaN 0.0 0.077 NaN NaN NaN NaN NaN
2 NaN NaN NaN 0.0 0.106 NaN NaN NaN NaN NaN
3 NaN NaN NaN 0.0 0.030 0.047 NaN NaN NaN NaN
4 NaN NaN NaN 0.0 0.143 0.084 0.0 0.0 0.09 NaN
N_183 N_184 N_185 N_186 N_187 N_188 N_189 N_190 N_191 N_192 \
0 NaN NaN NaN NaN NaN 0.69 NaN NaN NaN NaN
1 NaN NaN NaN NaN NaN 52.54 NaN NaN NaN NaN
2 NaN NaN NaN NaN NaN 0.25 NaN NaN NaN NaN
3 NaN NaN NaN NaN NaN 0.39 NaN 0.015 0.029 0.047
4 NaN NaN NaN NaN NaN 0.12 NaN 0.027 0.052 0.115
N_193 N_194 N_195 N_196 N_197 N_198 N_199 N_200 N_201 N_202 \
0 NaN 8.0 0.0 0.0 0.034 8.2 0.0 NaN NaN NaN
1 NaN 0.0 0.0 0.0 0.136 0.0 0.0 NaN NaN NaN
2 NaN 0.0 0.0 0.0 0.174 2.3 0.0 NaN NaN NaN
3 1926.0 96.0 0.0 0.0 0.054 10.0 0.0 0.0 NaN NaN
4 756.0 38.0 0.0 0.0 0.091 5.6 0.0 0.0 NaN NaN
N_203 N_204 N_205 N_206 N_207 N_208 N_209 N_210 N_211
0 NaN 0.02 0.0 NaN NaN 30.5 92.82 NaN 0.92
1 NaN 23.90 0.0 NaN NaN 0.0 2.41 NaN 3.31
2 NaN 0.12 0.0 NaN NaN 3.5 15.46 NaN 0.36
3 NaN 0.89 0.0 NaN NaN 3.3 86.35 NaN 0.20
4 NaN 1.13 0.0 0.0 NaN 41.6 93.22 NaN 0.54
3.1.4 EDA
# 查看数据缺失情况pd.set_option('display.max_rows', None)
((food.isnull().sum())/food.shape[0]).sort_values(ascending=False).map(lambda x:"{:.2%}".format(x))
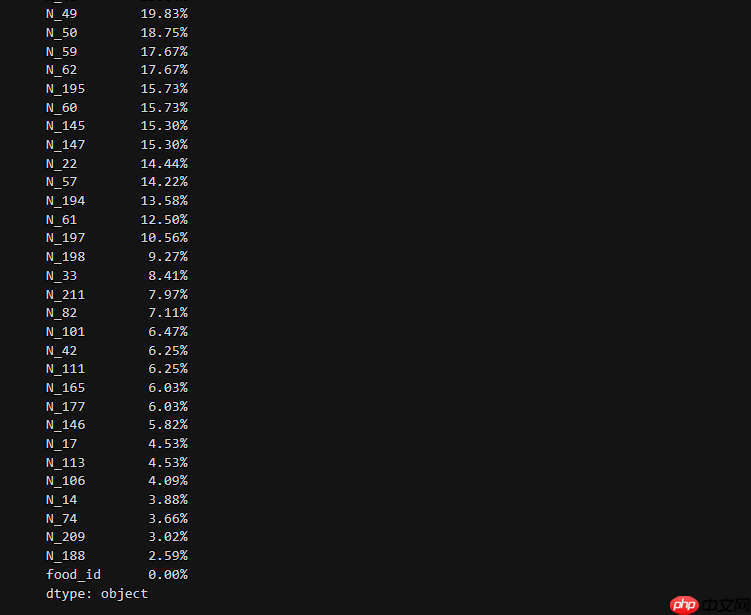
#只保留缺失率少于10%的列food=food[['N_198','N_33','N_211','N_82','N_101','N_42','N_111','N_165','N_177','N_146','N_17','N_113','N_106','N_14','N_74','N_209','N_188','food_id' ]] food.head(5)
N_198 N_33 N_211 N_82 N_101 N_42 N_111 N_165 N_177 N_146 \
0 8.2 0.157 0.92 27.0 0.481 23.0 79.0 6.0 0.076 0.126
1 0.0 1.099 3.31 279.0 3.637 598.0 713.0 3.0 0.077 1.197
2 2.3 0.272 0.36 36.0 0.766 299.0 744.0 26.0 0.106 0.125
3 10.0 0.078 0.20 10.0 0.600 124124.5 259.0 1.0 0.030 0.040
4 5.6 0.189 0.54 14.0 0.978 52.5 202.0 2.0 0.143 0.141
N_17 N_113 N_106 N_14 N_74 N_209 N_188 food_id
0 2.10 3.99 70.0 32.0 0.96 92.82 0.69 food_0
1 21.01 20.96 471.0 268.0 3.73 2.41 52.54 food_1
2 79.32 3.30 98.0 62.0 1.79 15.46 0.25 food_4
3 11.12 1.40 23.0 13.0 0.39 86.35 0.39 food_5
4 3.88 2.20 52.0 24.0 2.14 93.22 0.12 food_6
3.2 数据处理
3.2.1 目标编码
由于本题只有两个离散变量food_id 和disease_id ,而测试集中都是新的foodid
cat_list = ['disease']def stat(df, df_merge, group_by, agg):
group = df.groupby(group_by).agg(agg)
columns = [] for on, methods in agg.items(): for method in methods:
columns.append('{}_{}_{}'.format('_'.join(group_by), on, method))
group.columns = columns
group.reset_index(inplace=True)
df_merge = df_merge.merge(group, on=group_by, how='left') del (group)
gc.collect() return df_mergedef statis_feat(df_know, df_unknow,cat_list):
for f in tqdm(cat_list):
df_unknow = stat(df_know, df_unknow, [f], {'related': ['mean']}) return df_unknow
df_train = data[~data['related'].isnull()]
df_train = df_train.reset_index(drop=True)
df_test = data[data['related'].isnull()]
df_stas_feat = Nonekf = StratifiedKFold(n_splits=5, random_state=2020, shuffle=True)for train_index, val_index in kf.split(df_train, df_train['related']):
df_fold_train = df_train.iloc[train_index]
df_fold_val = df_train.iloc[val_index]
df_fold_val = statis_feat(df_fold_train, df_fold_val,cat_list)
df_stas_feat = pd.concat([df_stas_feat, df_fold_val], axis=0) del (df_fold_train) del (df_fold_val)
gc.collect()
df_test = statis_feat(df_train, df_test,cat_list)
data = pd.concat([df_stas_feat, df_test], axis=0)
data = data.reset_index(drop=True)del (df_stas_feat)del (df_train)del (df_test)
100%|██████████| 1/1 [00:00<00:00, 7.57it/s] 100%|██████████| 1/1 [00:00<00:00, 9.00it/s] 100%|██████████| 1/1 [00:00<00:00, 9.45it/s] 100%|██████████| 1/1 [00:00<00:00, 8.77it/s] 100%|██████████| 1/1 [00:00<00:00, 8.65it/s] 100%|██████████| 1/1 [00:00<00:00, 9.16it/s]
3.2.1 疾病特征处理
使用TruncatedSVD 的方法,对疾病特征进行降维,维度均为128
f_col = [col for col in disease_feature1.columns if 'F' in col]
disease_feature_1_ = disease_feature1.copy()from sklearn.feature_extraction.text import CountVectorizer,TfidfVectorizer,TfidfTransformer from sklearn.decomposition import TruncatedSVD, SparsePCA disease_feature_1_ = disease_feature_1_.fillna(0) decom=TruncatedSVD(n_components=128, n_iter = 20, random_state=2023) decom_x=decom.fit_transform(disease_feature_1_.iloc[:,1:]) decom_feas=pd.DataFrame(decom_x) decom_feas.columns=['disease1_svd_'+str(i) for i in range(decom_x.shape[1])]
disease_feature1 = disease_feature1[['disease_id']]for col in decom_feas:
disease_feature1[col] = decom_feas[col]
f_col = [col for col in disease_feature2.columns if 'F' in col]
disease_feature_2_ = disease_feature2.copy()from sklearn.feature_extraction.text import CountVectorizer,TfidfVectorizer,TfidfTransformer from sklearn.decomposition import TruncatedSVD, SparsePCA disease_feature_2_ = disease_feature_2_.fillna(0) decom=TruncatedSVD(n_components=128, n_iter = 20, random_state=2023) decom_x=decom.fit_transform(disease_feature_2_.iloc[:,1:]) decom_feas=pd.DataFrame(decom_x) decom_feas.columns=['disease2_svd_'+str(i) for i in range(decom_x.shape[1])]
disease_feature2 = disease_feature2[['disease_id']]for col in decom_feas:
disease_feature2[col] = decom_feas[col]
f_col = [col for col in disease_feature3.columns if 'F' in col]
disease_feature_3_ = disease_feature3.copy()from sklearn.feature_extraction.text import CountVectorizer,TfidfVectorizer,TfidfTransformer from sklearn.decomposition import TruncatedSVD, SparsePCA disease_feature_3_ = disease_feature_3_.fillna(0) decom=TruncatedSVD(n_components=128, n_iter = 20, random_state=2023) decom_x=decom.fit_transform(disease_feature_3_.iloc[:,1:]) decom_feas=pd.DataFrame(decom_x) decom_feas.columns=['disease3_svd_'+str(i) for i in range(decom_x.shape[1])]
disease_feature3 = disease_feature3[['disease_id']]for col in decom_feas:
disease_feature3[col] = decom_feas[col]
data = data.merge(food, on = 'food_id', how = 'left') data = data.merge(disease_feature1, on = 'disease_id', how = 'left') data = data.merge(disease_feature2, on = 'disease_id', how = 'left') data = data.merge(disease_feature3, on = 'disease_id', how = 'left') data.head()
food_id disease_id related food disease disease_related_mean N_198 \
0 food_0 disease_861 0.0 0 861 0.003521 8.2
1 food_0 disease_839 0.0 0 839 0.007299 8.2
2 food_0 disease_50 0.0 0 50 0.018382 8.2
3 food_0 disease_1370 0.0 0 1370 0.214286 8.2
4 food_0 disease_1015 0.0 0 1015 0.202749 8.2
N_33 N_211 N_82 N_101 N_42 N_111 N_165 N_177 N_146 N_17 N_113 \
0 0.157 0.92 27.0 0.481 23.0 79.0 6.0 0.076 0.126 2.1 3.99
1 0.157 0.92 27.0 0.481 23.0 79.0 6.0 0.076 0.126 2.1 3.99
2 0.157 0.92 27.0 0.481 23.0 79.0 6.0 0.076 0.126 2.1 3.99
3 0.157 0.92 27.0 0.481 23.0 79.0 6.0 0.076 0.126 2.1 3.99
4 0.157 0.92 27.0 0.481 23.0 79.0 6.0 0.076 0.126 2.1 3.99
N_106 N_14 N_74 N_209 N_188 disease1_svd_0 disease1_svd_1 \
0 70.0 32.0 0.96 92.82 0.69 NaN NaN
1 70.0 32.0 0.96 92.82 0.69 NaN NaN
2 70.0 32.0 0.96 92.82 0.69 NaN NaN
3 70.0 32.0 0.96 92.82 0.69 NaN NaN
4 70.0 32.0 0.96 92.82 0.69 NaN NaN
disease1_svd_2 disease1_svd_3 disease1_svd_4 disease1_svd_5 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_6 disease1_svd_7 disease1_svd_8 disease1_svd_9 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_10 disease1_svd_11 disease1_svd_12 disease1_svd_13 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_14 disease1_svd_15 disease1_svd_16 disease1_svd_17 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_18 disease1_svd_19 disease1_svd_20 disease1_svd_21 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_22 disease1_svd_23 disease1_svd_24 disease1_svd_25 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_26 disease1_svd_27 disease1_svd_28 disease1_svd_29 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_30 disease1_svd_31 disease1_svd_32 disease1_svd_33 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_34 disease1_svd_35 disease1_svd_36 disease1_svd_37 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_38 disease1_svd_39 disease1_svd_40 disease1_svd_41 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_42 disease1_svd_43 disease1_svd_44 disease1_svd_45 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_46 disease1_svd_47 disease1_svd_48 disease1_svd_49 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_50 disease1_svd_51 disease1_svd_52 disease1_svd_53 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_54 disease1_svd_55 disease1_svd_56 disease1_svd_57 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_58 disease1_svd_59 disease1_svd_60 disease1_svd_61 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_62 disease1_svd_63 disease1_svd_64 disease1_svd_65 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_66 disease1_svd_67 disease1_svd_68 disease1_svd_69 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_70 disease1_svd_71 disease1_svd_72 disease1_svd_73 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_74 disease1_svd_75 disease1_svd_76 disease1_svd_77 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_78 disease1_svd_79 disease1_svd_80 disease1_svd_81 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_82 disease1_svd_83 disease1_svd_84 disease1_svd_85 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_86 disease1_svd_87 disease1_svd_88 disease1_svd_89 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_90 disease1_svd_91 disease1_svd_92 disease1_svd_93 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_94 disease1_svd_95 disease1_svd_96 disease1_svd_97 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_98 disease1_svd_99 disease1_svd_100 disease1_svd_101 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_102 disease1_svd_103 disease1_svd_104 disease1_svd_105 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_106 disease1_svd_107 disease1_svd_108 disease1_svd_109 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_110 disease1_svd_111 disease1_svd_112 disease1_svd_113 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_114 disease1_svd_115 disease1_svd_116 disease1_svd_117 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_118 disease1_svd_119 disease1_svd_120 disease1_svd_121 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_122 disease1_svd_123 disease1_svd_124124 disease1_svd_125 \
0 NaN NaN NaN NaN
1 NaN NaN NaN NaN
2 NaN NaN NaN NaN
3 NaN NaN NaN NaN
4 NaN NaN NaN NaN
disease1_svd_126 disease1_svd_127 disease2_svd_0 disease2_svd_1 \
0 NaN NaN 21.207893 0.450860
1 NaN NaN 35.865549 -0.229259
2 NaN NaN 41.508018 -0.271734
3 NaN NaN 41.529790 -0.260051
4 NaN NaN 1.194324 0.016060
disease2_svd_2 disease2_svd_3 disease2_svd_4 disease2_svd_5 \
0 0.247976 -0.094030 -0.010093 -0.165738
1 -0.043399 -0.001870 -0.052036 -0.012363
2 -0.049130 -0.031626 -0.067875 -0.005537
3 -0.054846 -0.039315 -0.049941 0.002167
4 0.021213 -0.019584 0.053625 0.013038
disease2_svd_6 disease2_svd_7 disease2_svd_8 disease2_svd_9 \
0 0.063872 -0.106507 -0.206626 0.123055
1 -0.024822 -0.016792 -0.026163 -0.001283
2 -0.014704 -0.017518 -0.034036 -0.000631
3 -0.012543 -0.030178 -0.019744 -0.009011
4 0.009994 -0.015606 0.062566 -0.061401
disease2_svd_10 disease2_svd_11 disease2_svd_12 disease2_svd_13 \
0 -0.046291 0.103308 0.114410 -0.150404
1 -0.001989 0.002888 -0.007876 0.008097
2 -0.004023 0.013805 0.000356 -0.005335
3 -0.001326 0.012412445 0.004044 -0.010835
4 0.022027 -0.038624 -0.055178 -0.048019
disease2_svd_14 disease2_svd_15 disease2_svd_16 disease2_svd_17 \
0 0.008024 -0.075472 0.328313 -0.054091
1 0.001597 -0.000355 -0.000594 -0.002079
2 0.003229 0.000713 0.000098 -0.005014
3 -0.004046 0.019301 0.002939 -0.006936
4 0.759425 0.398878 0.111763 0.015837
disease2_svd_18 disease2_svd_19 disease2_svd_20 disease2_svd_21 \
0 0.210355 0.139096 -0.028611 0.080221
1 0.020368 -0.003079 0.021870 -0.008940
2 -0.005383 -0.005519 0.003013 -0.002217
3 -0.004579 -0.001534 -0.000085 0.007771
4 0.054869 -0.047739 0.036110 -0.022543
disease2_svd_22 disease2_svd_23 disease2_svd_24 disease2_svd_25 \
0 -0.151398 0.034177 0.041904 0.002051
1 0.004237 -0.004279 0.015770 0.001230
2 -0.009330 -0.005702 0.001625 -0.008246
3 -0.027475 -0.001232 0.001875 0.000077
4 0.029236 -0.030270 -0.006784 -0.003725
disease2_svd_26 disease2_svd_27 disease2_svd_28 disease2_svd_29 \
0 -0.131626 0.076600 -0.131167 0.006474
1 0.004035 -0.001987 0.010776 -0.003411
2 -0.001536 -0.006982 0.013521 0.000661
3 -0.008815 -0.020402 -0.001688 -0.008872
4 -0.032971 0.016045 0.015254 -0.008581
disease2_svd_30 disease2_svd_31 disease2_svd_32 disease2_svd_33 \
0 -0.097548 0.012412424 0.028256 0.006985
1 -0.009880 0.001792 -0.013636 -0.000586
2 -0.003444 0.004891 -0.000032 -0.000943
3 -0.005646 0.016212 0.013411 -0.018793
4 -0.012702 -0.011345 -0.010733 0.009206
disease2_svd_34 disease2_svd_35 disease2_svd_36 disease2_svd_37 \
0 0.010723 0.017358 -0.011925 -0.021033
1 0.011546 -0.014159 0.000428 -0.002775
2 -0.001430 0.004226 0.000832 0.002662
3 0.016397 -0.003017 -0.004065 0.003183
4 -0.009229 0.000618 0.005975 -0.003449
disease2_svd_38 disease2_svd_39 disease2_svd_40 disease2_svd_41 \
0 0.015393 0.006445 -0.019345 -0.033685
1 -0.001416 0.009633 0.003450 -0.003883
2 0.003511 0.000865 -0.001511 0.001598
3 -0.006690 0.018216 -0.004084 -0.009724
4 -0.007818 -0.009403 0.007591 0.002266
disease2_svd_42 disease2_svd_43 disease2_svd_44 disease2_svd_45 \
0 -0.030142 0.020834 0.023872 -0.025286
1 -0.002826 0.000251 0.009810 -0.008360
2 0.003186 -0.005701 -0.000564 0.000999
3 0.006243 -0.004513 -0.000231 0.000521
4 -0.004004 -0.002725 -0.003839 0.002820
disease2_svd_46 disease2_svd_47 disease2_svd_48 disease2_svd_49 \
0 0.003081 0.007100 0.019440 0.003406
1 -0.000637 0.003437 -0.002244 0.002247
2 0.000164 0.000142 0.002312 0.001523
3 -0.000797 0.003050 -0.001297 -0.004875
4 0.000835 -0.002778 0.000629 -0.002743
disease2_svd_50 disease2_svd_51 disease2_svd_52 disease2_svd_53 \
0 -0.015664 0.007384 0.004627 0.010140
1 -0.000411 0.000358 0.000566 0.002944
2 0.002301 -0.000096 -0.000341 0.000267
3 -0.003577 -0.001533 -0.001472 0.001390
4 -0.000550 0.000052 0.000554 -0.001329
disease2_svd_54 disease2_svd_55 disease2_svd_56 disease2_svd_57 \
0 0.016964 -0.001596 -0.000890 -0.002959
1 -0.000523 -0.000021 -0.000101 -0.000175
2 0.001574 -0.000061 0.000018 -0.000039
3 0.000372 -0.000034 -0.000062 -0.000605
4 0.000971 0.000060 0.000091 0.000130
disease2_svd_58 disease2_svd_59 disease2_svd_60 disease2_svd_61 \
0 0.004079 -0.001305 0.000426 0.000269
1 0.000425 0.000068 0.000147 0.000108
2 -0.000093 -0.000128 -0.000114 -0.000045
3 0.000263 -0.000107 0.000003 0.000030
4 -0.000430 -0.000074 -0.000154 -0.000046
disease2_svd_62 disease2_svd_63 disease2_svd_64 disease2_svd_65 \
0 0.000391 0.000193 1.491195e-04 -0.000022
1 -0.000043 -0.000021 1.726565e-05 -0.000090
2 0.000055 -0.000003 -8.250175e-06 0.000030
3 0.000036 0.000024 -8.291036e-07 -0.000040
4 0.000024 0.000015 -4.640532e-06 -0.000004
disease2_svd_66 disease2_svd_67 disease2_svd_68 disease2_svd_69 \
0 0.000075 -0.000328 -0.000104 -0.000818
1 -0.000079 0.000011 -0.000041 0.000142
2 0.000038 -0.000022 0.000025 -0.000064
3 -0.000080 0.000041 -0.000044 -0.000096
4 0.000006 0.000035 0.000012 -0.000102
disease2_svd_70 disease2_svd_71 disease2_svd_72 disease2_svd_73 \
0 -0.000536 0.000425 0.000675 0.000525
1 -0.000004 0.000041 -0.000045 -0.000070
2 -0.000052 -0.000010 0.000063 -0.000024
3 -0.000057 0.000017 0.000058 -0.000022
4 -0.000041 -0.000042 0.000003 0.000004
disease2_svd_74 disease2_svd_75 disease2_svd_76 disease2_svd_77 \
0 -0.000909 0.000450 -0.000754 0.002798
1 -0.000194 0.000516 0.000210 0.000677
2 0.000188 -0.000111 -0.000098 -0.000364
3 -0.000132 0.000107 0.000208 0.000220
4 0.000252 -0.000294 -0.000062 -0.000560
disease2_svd_78 disease2_svd_79 disease2_svd_80 disease2_svd_81 \
0 -0.001876 -0.003088 -0.002775 -0.001081
1 -0.000440 -0.000186 -0.000202 -0.000044
2 -0.000028 0.000122 -0.000164 0.000070
3 -0.000585 -0.000699 -0.000664 0.000296
4 0.000023 -0.000038 -0.000145 0.000341
disease2_svd_82 disease2_svd_83 disease2_svd_84 disease2_svd_85 \
0 0.000486 0.004900 0.000529 0.004128
1 -0.000397 0.001779 0.000118 -0.000687
2 -0.000014 -0.000859 -0.000266 0.000222
3 -0.000517 0.000028 0.000650 -0.000914
4 0.000053 -0.001102 0.000195 -0.000488
disease2_svd_86 disease2_svd_87 disease2_svd_88 disease2_svd_89 \
0 0.009350 0.002341 -0.007186 -0.009234
1 0.000381 0.000223 0.000390 -0.000753
2 0.000684 -0.000063 -0.000192 -0.000442
3 -0.000190 0.000124124 -0.001585 -0.000599
4 -0.000224 0.000328 -0.000370 0.000285
disease2_svd_90 disease2_svd_91 disease2_svd_92 disease2_svd_93 \
0 -0.007480 0.001355 0.003528 -0.017621
1 -0.000451 -0.002448 -0.001939 -0.001105
2 0.000511 0.000777 0.000287 -0.000420
3 -0.000961 0.000682 0.000873 -0.000953
4 0.001096 -0.000158 0.001338 0.000476
disease2_svd_94 disease2_svd_95 disease2_svd_96 disease2_svd_97 \
0 0.005953 0.010327 0.004300 0.024545
1 -0.000197 -0.001683 0.004916 0.001206
2 0.000969 0.001567 0.001200 -0.000256
3 0.001415 -0.004218 0.004151 0.003551
4 0.002296 0.000573 0.001374 -0.001213
disease2_svd_98 disease2_svd_99 disease2_svd_100 disease2_svd_101 \
0 0.016885 0.013065 -0.004008 0.020067
1 -0.000385 -0.004373 0.004536 0.001937
2 0.000491 -0.000133 -0.000430 -0.001268
3 0.000414 -0.001865 -0.000427 -0.000561
4 -0.000561 0.000960 -0.000290 -0.000561
disease2_svd_102 disease2_svd_103 disease2_svd_104 disease2_svd_105 \
0 -0.027640 -0.020863 0.002466 -0.009659
1 -0.003012 -0.001530 0.002150 0.001011
2 -0.002388 -0.000176 -0.002063 -0.002062
3 0.000712 0.000712 -0.002766 -0.002378
4 -0.001062 -0.001659 0.000637 -0.004378
disease2_svd_106 disease2_svd_107 disease2_svd_108 disease2_svd_109 \
0 -0.004832 -0.006345 0.007009 0.018090
1 -0.001764 0.003546 -0.001815 0.002938
2 0.001144 0.001614 0.001952 0.002008
3 0.000731 -0.000705 0.004565 -0.001887
4 -0.002726 0.002012 0.000543 0.001945
disease2_svd_110 disease2_svd_111 disease2_svd_112 disease2_svd_113 \
0 -0.005579 -0.001987 0.037768 0.012893
1 -0.003030 -0.009093 -0.002076 -0.004467
2 0.000060 0.001320 -0.000544 -0.000907
3 -0.005995 0.003522 0.002315 -0.002299
4 0.005084 0.001443 0.000259 -0.000784
disease2_svd_114 disease2_svd_115 disease2_svd_116 disease2_svd_117 \
0 0.002925 0.044773 -0.038541 0.005572
1 0.007585 0.001704 -0.000898 -0.000604
2 0.003609 0.001488 -0.000267 -0.002821
3 0.004152 -0.007743 0.002656 -0.005594
4 0.001073 -0.002402 0.005187 0.002465
disease2_svd_118 disease2_svd_119 disease2_svd_120 disease2_svd_121 \
0 0.015253 0.031836 -0.001107 0.059203
1 -0.002589 0.007437 0.006040 0.009314
2 0.000381 -0.003625 0.001080 -0.003746
3 -0.001302 0.003646 -0.001710 0.006324
4 -0.000312 -0.000665 0.002359 0.003525
disease2_svd_122 disease2_svd_123 disease2_svd_124124 disease2_svd_125 \
0 0.009907 -0.044329 0.045791 -0.019859
1 0.007324 -0.008239 -0.007265 -0.007121
2 -0.000740 0.004468 0.000185 0.005591
3 0.006646 -0.006136 0.003705 0.010991
4 0.000040 0.003171 -0.006081 -0.004884
disease2_svd_126 disease2_svd_127 disease3_svd_0 disease3_svd_1 \
0 -0.008714 -0.057113 1.081144 -0.096631
1 -0.002937 -0.000852 1.817120 -1.784037
2 -0.001899 0.001580 1.189615 -0.109329
3 -0.006933 0.001896 1.070765 -0.808722
4 0.008354 -0.002090 1.286808 -0.267558
disease3_svd_2 disease3_svd_3 disease3_svd_4 disease3_svd_5 \
0 0.610973 0.214094 0.285724 0.037680
1 -0.973115 -0.322312 0.991550 2.330509
2 0.002665 0.190674 0.706729 -0.526051
3 -0.295265 -0.377572 0.224404 0.624164
4 0.293177 0.179919 0.753527 -0.548435
disease3_svd_6 disease3_svd_7 disease3_svd_8 disease3_svd_9 \
0 2.024824 0.396048 -0.701736 -0.903993
1 -0.176316 -0.130993 -0.397610 -0.210186
2 0.151012 1.535668 1.386908 -0.136352
3 -0.171772 -0.033262 -0.001275 -0.288270
4 -0.262182 0.455970 0.503655 -0.740368
disease3_svd_10 disease3_svd_11 disease3_svd_12 disease3_svd_13 \
0 -0.163829 -0.665074 -0.146170 0.292248
1 0.532973 0.323650 0.015021 0.053810
2 0.122367 -0.631401 0.126397 -0.276903
3 -0.386495 0.179418 0.970317 -0.800248
4 -0.459004 -0.473660 0.816256 -0.538978
disease3_svd_14 disease3_svd_15 disease3_svd_16 disease3_svd_17 \
0 -0.264666 0.232114 0.142807 0.018816
1 0.220851 -0.044089 -0.129121 0.357842
2 0.329921 -0.058434 0.150027 0.081792
3 -1.032255 0.141866 -0.130448 0.211681
4 1.083409 0.272414 -0.283723 -0.921948
disease3_svd_18 disease3_svd_19 disease3_svd_20 disease3_svd_21 \
0 0.006140 0.215335 0.123988 -0.007711
1 -0.118835 0.533157 0.197675 0.033118
2 -0.109353 0.098505 -0.615293 -0.061401
3 -0.161183 -0.133450 0.103317 -0.330761
4 0.089385 -0.305596 -0.141462 0.209232
disease3_svd_22 disease3_svd_23 disease3_svd_24 disease3_svd_25 \
0 0.503743 0.082692 -0.192049 0.049991
1 0.212645 0.058697 -0.138553 -0.074376
2 0.054187 0.171404 0.067380 -0.178878
3 -0.286104 -0.109326 0.120395 0.188776
4 -0.102136 0.361745 -0.073097 0.247010
disease3_svd_26 disease3_svd_27 disease3_svd_28 disease3_svd_29 \
0 0.284193 -0.419728 -0.265826 0.219807
1 -0.076036 0.418851 -0.044005 -0.103922
2 0.316629 0.142819 -0.221195 -0.169382
3 0.892057 -0.053633 0.402967 0.386931
4 -0.215279 0.622676 -0.036221 -0.100733
disease3_svd_30 disease3_svd_31 disease3_svd_32 disease3_svd_33 \
0 -0.083213 0.365347 0.014522 0.270022
1 0.011587 -0.067979 -0.088224 -0.190870
2 -0.276687 0.346723 -0.050159 -0.171241242
3 -0.284458 -0.034337 0.094869 -0.405306
4 -0.095383 0.252611 0.112919 -0.032485
disease3_svd_34 disease3_svd_35 disease3_svd_36 disease3_svd_37 \
0 -0.100975 -0.027756 -0.005296 -0.384114
1 0.234372 0.014150 0.295512 0.208233
2 -0.080506 -0.118671 0.018256 0.047224
3 -0.019650 0.119014 -0.220812 -0.157350
4 -0.269291 0.179181 -0.179328 -0.041581
disease3_svd_38 disease3_svd_39 disease3_svd_40 disease3_svd_41 \
0 -0.131914 -0.386147 0.037262 0.123429
1 -0.002893 0.028525 0.142613 -0.235298
2 -0.304223 -0.152287 0.122938 -0.058284
3 0.113438 0.337078 -0.105879 -0.191973
4 -0.208619 -0.143577 -0.258412 -0.121063
disease3_svd_42 disease3_svd_43 disease3_svd_44 disease3_svd_45 \
0 -0.193423 0.233849 -0.135330 0.132876
1 0.071875 0.035765 -0.018080 0.006652
2 -0.407862 -0.010772 -0.175505 0.132974
3 0.182164 0.150012 -0.002031 0.214086
4 -0.410299 0.081461 -0.118664 0.432868
disease3_svd_46 disease3_svd_47 disease3_svd_48 disease3_svd_49 \
0 -0.204344 0.048369 -0.094278 -0.219713
1 0.144174 0.107761 0.102500 -0.028223
2 0.031009 0.221593 -0.054744 -0.009640
3 -0.001215 0.217673 -0.046450 -0.045357
4 0.512245 -0.090577 0.357983 -0.083231
disease3_svd_50 disease3_svd_51 disease3_svd_52 disease3_svd_53 \
0 -0.040830 -0.112828 -0.003695 -0.135492
1 -0.166622 -0.227230 0.059775 -0.054151
2 -0.070451 -0.264516 0.110601 0.291928
3 0.213626 -0.012232 0.095100 0.030139
4 0.062759 0.106986 0.043856 -0.294729
disease3_svd_54 disease3_svd_55 disease3_svd_56 disease3_svd_57 \
0 0.027470 -0.069589 -0.110494 0.046717
1 0.143829 0.048532 0.072578 -0.095288
2 0.102618 -0.166557 -0.021385 0.064398
3 0.068430 -0.192951 -0.114827 0.035296
4 -0.021793 0.070910 -0.007633 -0.070904
disease3_svd_58 disease3_svd_59 disease3_svd_60 disease3_svd_61 \
0 -0.214439 0.046134 0.134354 -0.104612
1 -0.048502 0.015662 0.030757 -0.050883
2 0.245133 -0.163323 -0.205366 -0.078624
3 -0.216153 0.158659 0.111714 -0.154277
4 0.097167 0.162714 0.052339 0.102275
disease3_svd_62 disease3_svd_63 disease3_svd_64 disease3_svd_65 \
0 -0.104119 -0.080847 0.155709 0.017000
1 0.147684 0.008403 -0.135227 -0.081102
2 -0.113051 -0.086653 0.057236 0.089719
3 -0.135024 0.234255 -0.090858 0.091808
4 0.007705 -0.093869 -0.008447 0.046357
disease3_svd_66 disease3_svd_67 disease3_svd_68 disease3_svd_69 \
0 -0.169362 0.020168 0.120298 0.094487
1 -0.040909 -0.055877 0.008944 0.042602
2 0.022555 0.033048 0.041558 -0.154484
3 0.030877 -0.000725 -0.186567 -0.036827
4 0.154401 -0.124124018 -0.049133 0.308927
disease3_svd_70 disease3_svd_71 disease3_svd_72 disease3_svd_73 \
0 -0.117866 0.041941 -0.236982 -0.036641
1 -0.086106 -0.070377 -0.061552 0.005882
2 -0.283011 -0.139989 -0.030849 -0.023703
3 -0.024478 0.017350 -0.110065 -0.012707
4 -0.101389 0.107464 0.126912 -0.087374
disease3_svd_74 disease3_svd_75 disease3_svd_76 disease3_svd_77 \
0 0.017555 0.030866 0.045257 0.070255
1 -0.013598 0.051109 -0.037114 0.007101
2 -0.195976 0.047442 0.177886 -0.031600
3 -0.191022 0.007126 -0.027630 0.049433
4 0.120835 -0.130838 -0.116480 -0.070292
disease3_svd_78 disease3_svd_79 disease3_svd_80 disease3_svd_81 \
0 -0.035509 0.031680 -0.041112 -0.003302
1 -0.044109 0.004390 0.032941 -0.026301
2 -0.045078 0.172449 -0.196692 -0.226955
3 0.232110 -0.107700 0.023428 -0.015442
4 -0.049393 0.028696 0.104959 0.181812
disease3_svd_82 disease3_svd_83 disease3_svd_84 disease3_svd_85 \
0 -0.026433 0.150842 -0.117318 -0.081397
1 -0.014711 0.021407 0.017150 0.007859
2 -0.091421 0.116724 0.075902 0.198356
3 -0.073996 -0.168445 -0.069636 0.062691
4 0.097010 -0.075481 0.084535 -0.122547
disease3_svd_86 disease3_svd_87 disease3_svd_88 disease3_svd_89 \
0 0.013877 -0.030950 0.035688 -0.022059
1 -0.012711 -0.081762 0.013885 -0.030356
2 0.069463 -0.098914 -0.074465 0.018325
3 0.074576 0.089530 -0.082161 -0.074518
4 0.046716 0.049885 -0.094828 0.062870
disease3_svd_90 disease3_svd_91 disease3_svd_92 disease3_svd_93 \
0 -0.015251 -0.046003 -0.003584 0.058973
1 -0.024276 -0.090971 0.031763 -0.072141
2 0.013663 0.044086 0.006720 -0.092438
3 -0.042437 0.031088 -0.155015 0.019656
4 -0.062986 0.030545 0.001237 -0.047724
disease3_svd_94 disease3_svd_95 disease3_svd_96 disease3_svd_97 \
0 0.116162 -0.102288 -0.026451 -0.065339
1 0.062488 0.036716 -0.080577 -0.054123
2 0.106865 0.042334 -0.106831 0.047141
3 0.039321 -0.055407 -0.063896 -0.093685
4 -0.022991 0.004514 0.055843 0.048817
disease3_svd_98 disease3_svd_99 disease3_svd_100 disease3_svd_101 \
0 -0.016272 -0.020509 0.050898 -0.088068
1 0.011628 -0.033164 0.075565 0.054723
2 -0.052669 0.032439 -0.020543 -0.067151
3 0.226617 0.113530 -0.045031 -0.174211
4 0.044805 0.058769 -0.070773 0.088568
disease3_svd_102 disease3_svd_103 disease3_svd_104 disease3_svd_105 \
0 0.030054 -0.022857 0.045037 -0.100831
1 0.017667 0.054461 0.015337 -0.044796
2 0.024235 -0.023867 -0.022965 0.079472
3 -0.128222 -0.114841 -0.031775 0.003285
4 0.020527 -0.016702 0.049144 -0.057848
disease3_svd_106 disease3_svd_107 disease3_svd_108 disease3_svd_109 \
0 0.004140 0.012740 -0.070383 0.056177
1 -0.028650 -0.086811 -0.008269 0.086882
2 -0.012705 0.022978 0.045083 -0.016602
3 -0.002169 -0.108374 0.002132 0.015701
4 -0.151351 -0.153255 0.007800 0.052151
disease3_svd_110 disease3_svd_111 disease3_svd_112 disease3_svd_113 \
0 -0.029545 -0.034183 0.027649 0.003119
1 -0.057133 0.054998 -0.053585 -0.027943
2 0.069385 -0.034710 -0.012632 -0.055165
3 -0.025789 0.049046 -0.074700 0.020771
4 0.053366 0.039268 -0.006772 0.077348
disease3_svd_114 disease3_svd_115 disease3_svd_116 disease3_svd_117 \
0 0.028489 -0.058005 0.060475 0.159239
1 -0.031830 0.060304 0.009329 -0.003838
2 -0.023180 -0.105940 -0.096701 -0.139067
3 -0.000206 0.066936 0.028114 -0.094354
4 -0.028753 -0.118866 0.028853 -0.078241
disease3_svd_118 disease3_svd_119 disease3_svd_120 disease3_svd_121 \
0 0.003554 -0.050520 -0.049823 -0.017363
1 -0.004057 0.011732 -0.012960 -0.005264
2 0.082889 -0.008238 -0.058866 0.072690
3 -0.114083 0.041070 -0.060834 0.031545
4 -0.035130 -0.033633 -0.015799 -0.042921
disease3_svd_122 disease3_svd_123 disease3_svd_124124 disease3_svd_125 \
0 -0.116130 0.085801 0.072854 0.120381
1 0.024481 -0.021813 -0.020103 0.079306
2 -0.040983 0.001735 -0.031011 0.012370
3 0.020605 0.045850 0.060787 -0.054691
4 0.035031 0.071032 -0.097231 0.081445
disease3_svd_126 disease3_svd_127
0 0.033087 -0.025249
1 0.056902 -0.012589
2 -0.016576 0.062942
3 -0.081137 -0.041880
4 -0.014732 0.010713
3.2.3 交叉特征
这里按特征重要性选取靠前的部分特征进行交叉
topn = ['N_33', 'N_198', 'N_74','N_188','N_82','N_42','N_111','disease','food']for i in range(len(topn)): for j in range(i + 1, len(topn)):
data[f'{topn[i]}+{topn[j]}'] = data[topn[i]] + data[topn[j]]
data[f'{topn[i]}-{topn[j]}'] = data[topn[i]] - data[topn[j]]
data[f'{topn[i]}*{topn[j]}'] = data[topn[i]] * data[topn[j]]
data[f'{topn[i]}/{topn[j]}'] = data[topn[i]] / (data[topn[j]]+1e-5)
drop_cols = ['disease_id', 'food_id', 'related']
3.2.3 特征筛选
去除掉只有单一取值的特征
for f in data.columns: if data[f].nunique() < 2:
drop_cols.append(f)
test_df = data[data["related"].isnull() == True].copy().reset_index(drop=True) train_df = data[~data["related"].isnull() == True].copy().reset_index(drop=True)
feature_name = [f for f in train_df.columns if f not in drop_cols] X_train = train_df[feature_name].reset_index(drop=True) X_test = test_df[feature_name].reset_index(drop=True) y = train_df['related'].reset_index(drop=True)print(len(feature_name))print(feature_name)
548 ['food', 'disease', 'disease_related_mean', 'N_198', 'N_33', 'N_211', 'N_82', 'N_101', 'N_42', 'N_111', 'N_165', 'N_177', 'N_146', 'N_17', 'N_113', 'N_106', 'N_14', 'N_74', 'N_209', 'N_188', 'disease1_svd_0', 'disease1_svd_1', 'disease1_svd_2', 'disease1_svd_3', 'disease1_svd_4', 'disease1_svd_5', 'disease1_svd_6', 'disease1_svd_7', 'disease1_svd_8', 'disease1_svd_9', 'disease1_svd_10', 'disease1_svd_11', 'disease1_svd_12', 'disease1_svd_13', 'disease1_svd_14', 'disease1_svd_15', 'disease1_svd_16', 'disease1_svd_17', 'disease1_svd_18', 'disease1_svd_19', 'disease1_svd_20', 'disease1_svd_21', 'disease1_svd_22', 'disease1_svd_23', 'disease1_svd_24', 'disease1_svd_25', 'disease1_svd_26', 'disease1_svd_27', 'disease1_svd_28', 'disease1_svd_29', 'disease1_svd_30', 'disease1_svd_31', 'disease1_svd_32', 'disease1_svd_33', 'disease1_svd_34', 'disease1_svd_35', 'disease1_svd_36', 'disease1_svd_37', 'disease1_svd_38', 'disease1_svd_39', 'disease1_svd_40', 'disease1_svd_41', 'disease1_svd_42', 'd
print(test_df.shape)
(47212, 551)
3.2.3 模型训练
本次仅使用lightgbm模型来训练。
train_pred = {}
test_pred = {}
seeds = [2]
num_model_seed = 1oof = np.zeros(X_train.shape[0])
prediction = np.zeros(X_test.shape[0])
feat_imp_df = pd.DataFrame({'feats': feature_name, 'imp': 0})
parameters = { 'learning_rate': 0.05, 'boosting_type': 'gbdt', 'objective': 'binary', 'metric': 'auc', 'num_leaves': 63, 'feature_fraction': 0.8, 'bagging_fraction': 0.8, 'bagging_freq': 5, 'seed': 2022, 'bagging_seed': 1, 'feature_fraction_seed': 7, 'min_data_in_leaf': 20, 'verbose': -1,
'n_jobs':8,
}
fold = 5for model_seed in range(num_model_seed): print(seeds[model_seed],"--------------------------------------------------------------------------------------------")
oof_cat = np.zeros(X_train.shape[0])
prediction_cat = np.zeros(X_test.shape[0])
skf = StratifiedKFold(n_splits=fold, random_state=seeds[model_seed], shuffle=True) for index, (train_index, test_index) in enumerate(skf.split(X_train, y)):
train_x, test_x, train_y, test_y = X_train[feature_name].iloc[train_index], X_train[feature_name].iloc[test_index], y.iloc[train_index], y.iloc[test_index]
dtrain = lgb.Dataset(train_x, label=train_y)
dval = lgb.Dataset(test_x, label=test_y)
lgb_model = lgb.train(
parameters,
dtrain,
num_boost_round=10000,
valid_sets=[dval],
early_stopping_rounds=100,
verbose_eval=100, )
oof_cat[test_index] += lgb_model.predict(test_x,num_iteration=lgb_model.best_iteration)
prediction_cat += lgb_model.predict(X_test,num_iteration=lgb_model.best_iteration) / fold
feat_imp_df['imp'] += lgb_model.feature_importance() del train_x del test_x del train_y del test_y del lgb_model
oof += oof_cat / num_model_seed
prediction += prediction_cat / num_model_seed
gc.collect()
3.2.4 结果可视化
train_pred['lgb'] = oof test_pred['lgb'] = prediction
print("lgb train auc: ", roc_auc_score(y, train_pred['lgb']))
lgb train auc: 0.9778226537246766
scores = []; thresholds = []
best_score = 0; best_threshold = 0for threshold in np.arange(0.1,0.9,0.01): print(f'{threshold:.02f}, ',end='')
preds = (train_pred['lgb'].reshape((-1)) > threshold).astype('int')
m = f1_score(y.values.reshape((-1)), preds, average='binary')
scores.append(m)
thresholds.append(threshold) if m>best_score:
best_score = m
best_threshold = threshold
0.10, 0.11, 0.12, 0.13, 0.14, 0.15, 0.16, 0.17, 0.18, 0.19, 0.20, 0.21, 0.22, 0.23, 0.24, 0.25, 0.26, 0.27, 0.28, 0.29, 0.30, 0.31, 0.32, 0.33, 0.34, 0.35, 0.36, 0.37, 0.38, 0.39, 0.40, 0.41, 0.42, 0.43, 0.44, 0.45, 0.46, 0.47, 0.48, 0.49, 0.50, 0.51, 0.52, 0.53, 0.54, 0.55, 0.56, 0.57, 0.58, 0.59, 0.60, 0.61, 0.62, 0.63, 0.64, 0.65, 0.66, 0.67, 0.68, 0.69, 0.70, 0.71, 0.72, 0.73, 0.74, 0.75, 0.76, 0.77, 0.78, 0.79, 0.80, 0.81, 0.82, 0.83, 0.84, 0.85, 0.86, 0.87, 0.88, 0.89,
import matplotlib.pyplot as plt# PLOT THRESHOLD VS. F1_SCOREplt.figure(figsize=(20,5))
plt.plot(thresholds,scores,'-o',color='blue')
plt.scatter([best_threshold], [best_score], color='blue', s=300, alpha=1)
plt.xlabel('Threshold',size=14)
plt.ylabel('Validation F1 Score',size=14)
plt.title(f'Threshold vs. F1_Score with Best F1_Score = {best_score:.3f} at Best Threshold = {best_threshold:.3}',size=18)
plt.show()
auc = roc_auc_score(y, train_pred['lgb']) f1 = best_scoreprint((auc + f1) / 2)
0.8939347040505519
3.2.5 生成提交结果
控制1的个数为4100个左右,最终结果:
# label=[1 if x >= 0.265+0.235 else 0 for x in prediction+0.235]# np.sum(label)label=[1 if x >= 0.26+0.24 else 0 for x in prediction+0.25] np.sum(label)
4032
preliminary_a_submit_sample['related_prob'] = prediction+0.25
preliminary_a_submit_sample.to_csv('submit.csv', index=False)
四、项目总结
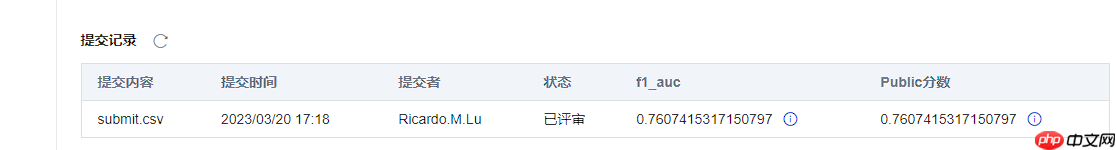
4.1 提交结果